¿Cómo hacer Flipbooks?
Con flipbookr y xaringan
Carlos A. Ar.
Última edición 06 de septiembre del 2021
¿Qué es un flipbook?
- Es una forma de presentar información de manera paulatina.
¿Qué es un flipbook?
Es una forma de presentar información de manera paulatina.
Los datos así presentados pueden:
¿Qué es un flipbook?
Es una forma de presentar información de manera paulatina.
Los datos así presentados pueden:
- Genearar ideas de información más eficiente que al ver una consola de código.
¿Qué es un flipbook?
Es una forma de presentar información de manera paulatina.
Los datos así presentados pueden:
Genearar ideas de información más eficiente que al ver una consola de código.
Dar a entender los cambios que se reflejan con el código, ya sea:
¿Qué es un flipbook?
Es una forma de presentar información de manera paulatina.
Los datos así presentados pueden:
Genearar ideas de información más eficiente que al ver una consola de código.
Dar a entender los cambios que se reflejan con el código, ya sea:
Manejo de datos.
Visualización de datos.
Análisis de datos.
¿Qué es un flipbook?
Es una forma de presentar información de manera paulatina.
Los datos así presentados pueden:
Genearar ideas de información más eficiente que al ver una consola de código.
Dar a entender los cambios que se reflejan con el código, ya sea:
Manejo de datos.
Visualización de datos.
Análisis de datos.
- Salvar a tu audiencia de un desmayo por aburrimiento.
¿Qué es un flipbook?
Es una forma de presentar información de manera paulatina.
Los datos así presentados pueden:
Genearar ideas de información más eficiente que al ver una consola de código.
Dar a entender los cambios que se reflejan con el código, ya sea:
Manejo de datos.
Visualización de datos.
Análisis de datos.
- Salvar a tu audiencia de un desmayo por aburrimiento.
Un paso a la vez.
¿Qué es flipbookr?
Un paquete de R que sirve para hacer un flipbook.
La maravillosa herramineta que permite convertir...
¿Qué es flipbookr?
Un paquete de R que sirve para hacer un flipbook.
La maravillosa herramineta que permite convertir...
Esto

¿Qué es flipbookr?
Un paquete de R que sirve para hacer un flipbook.
La maravillosa herramineta que permite convertir...
Esto

En esto
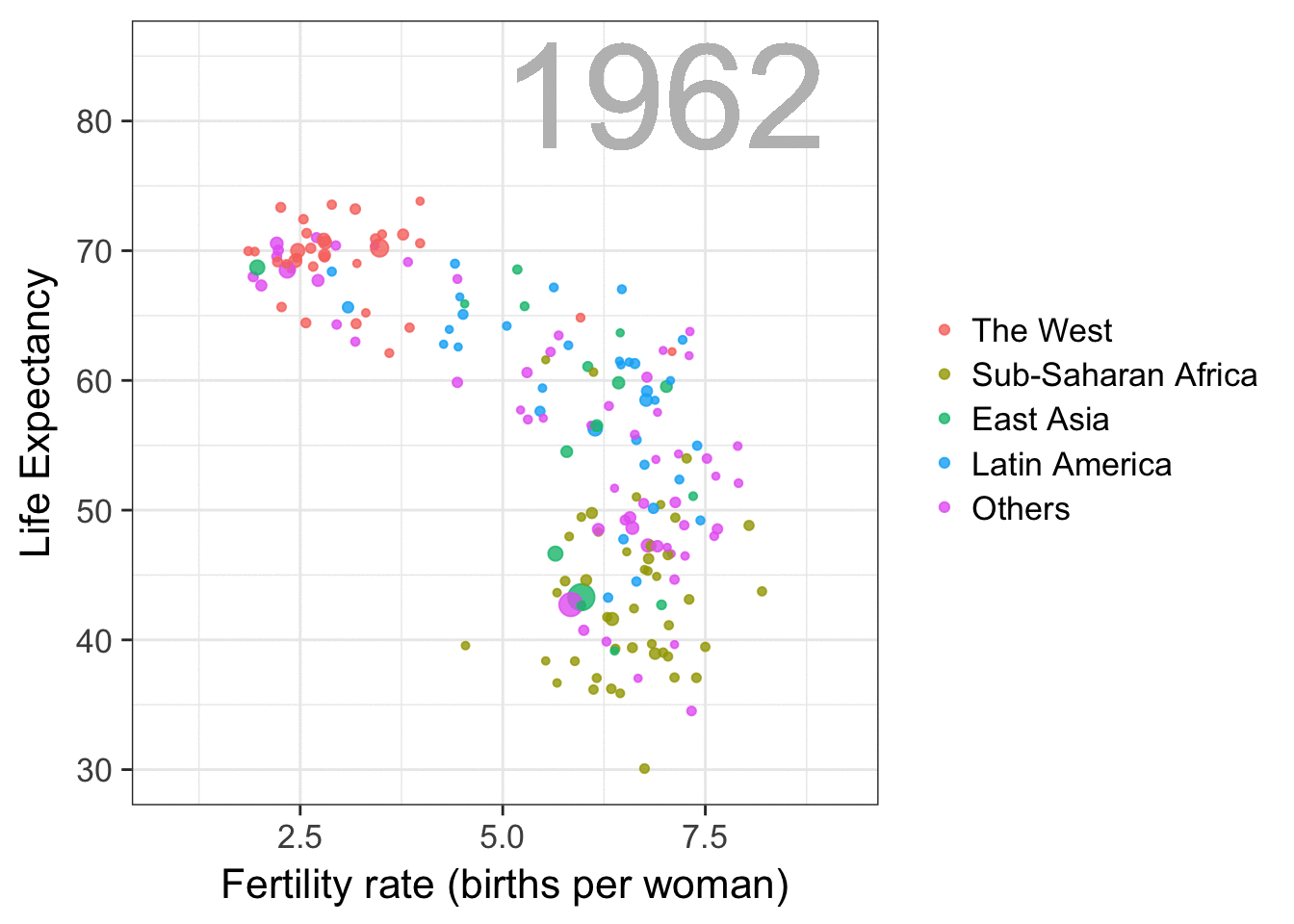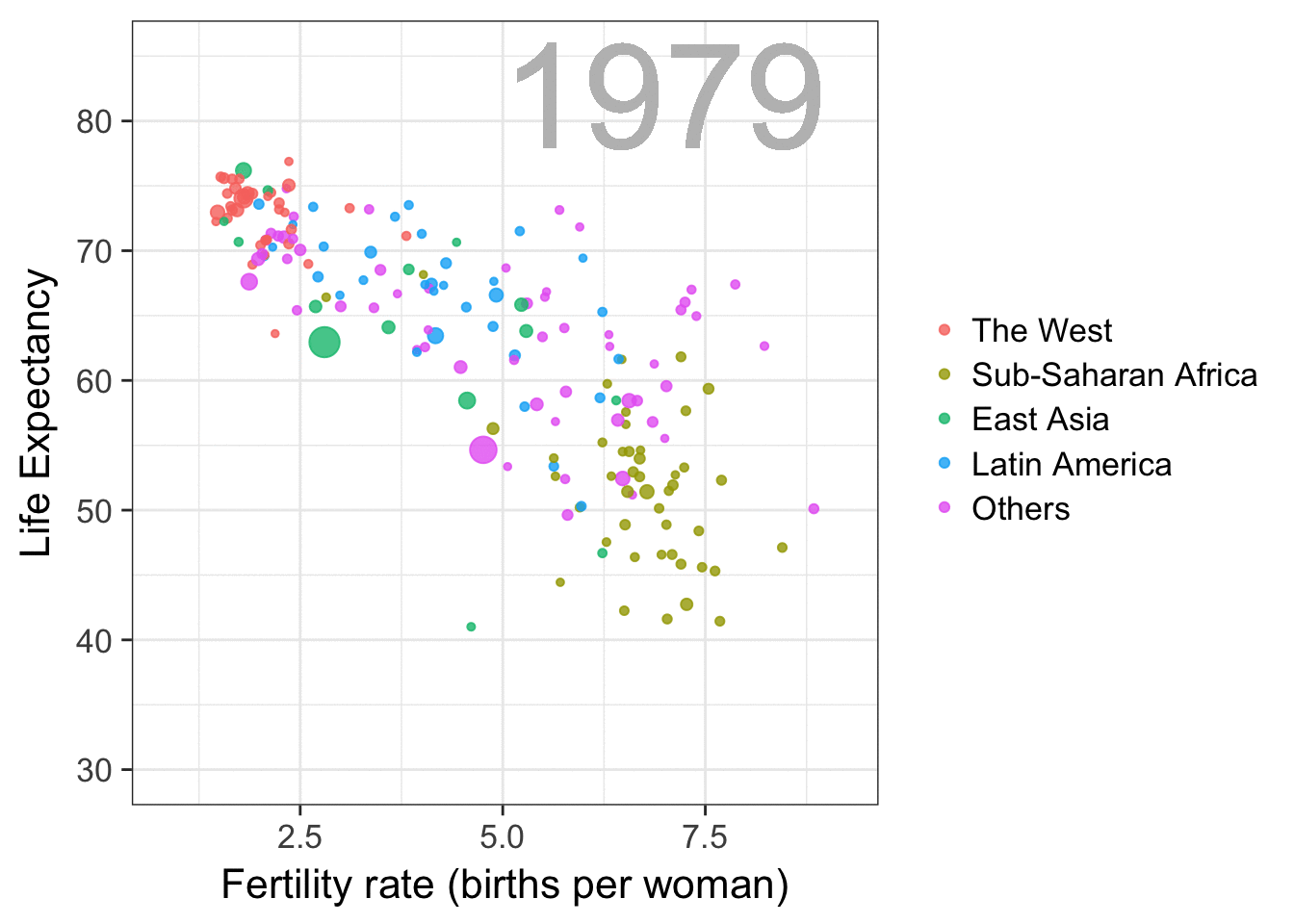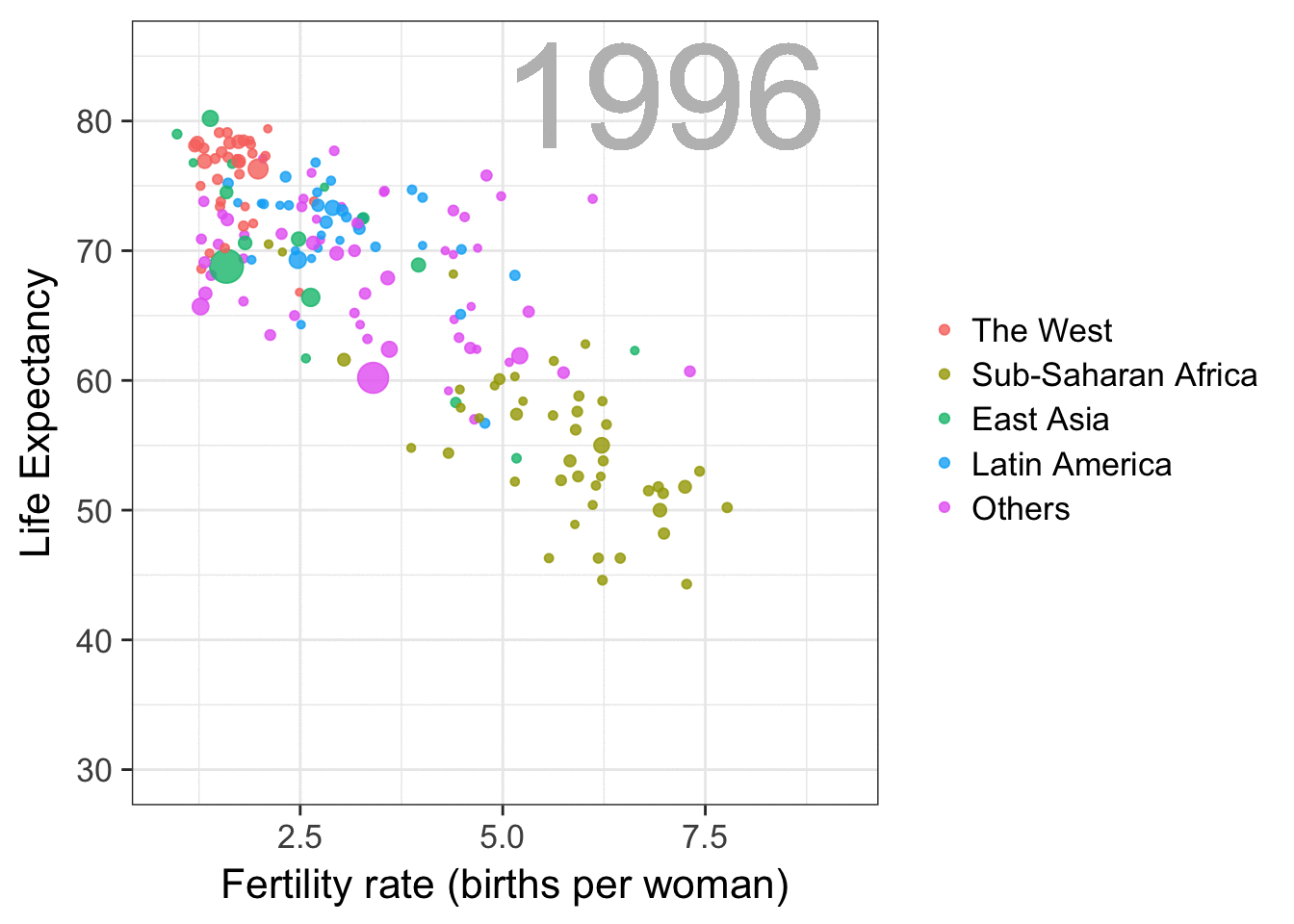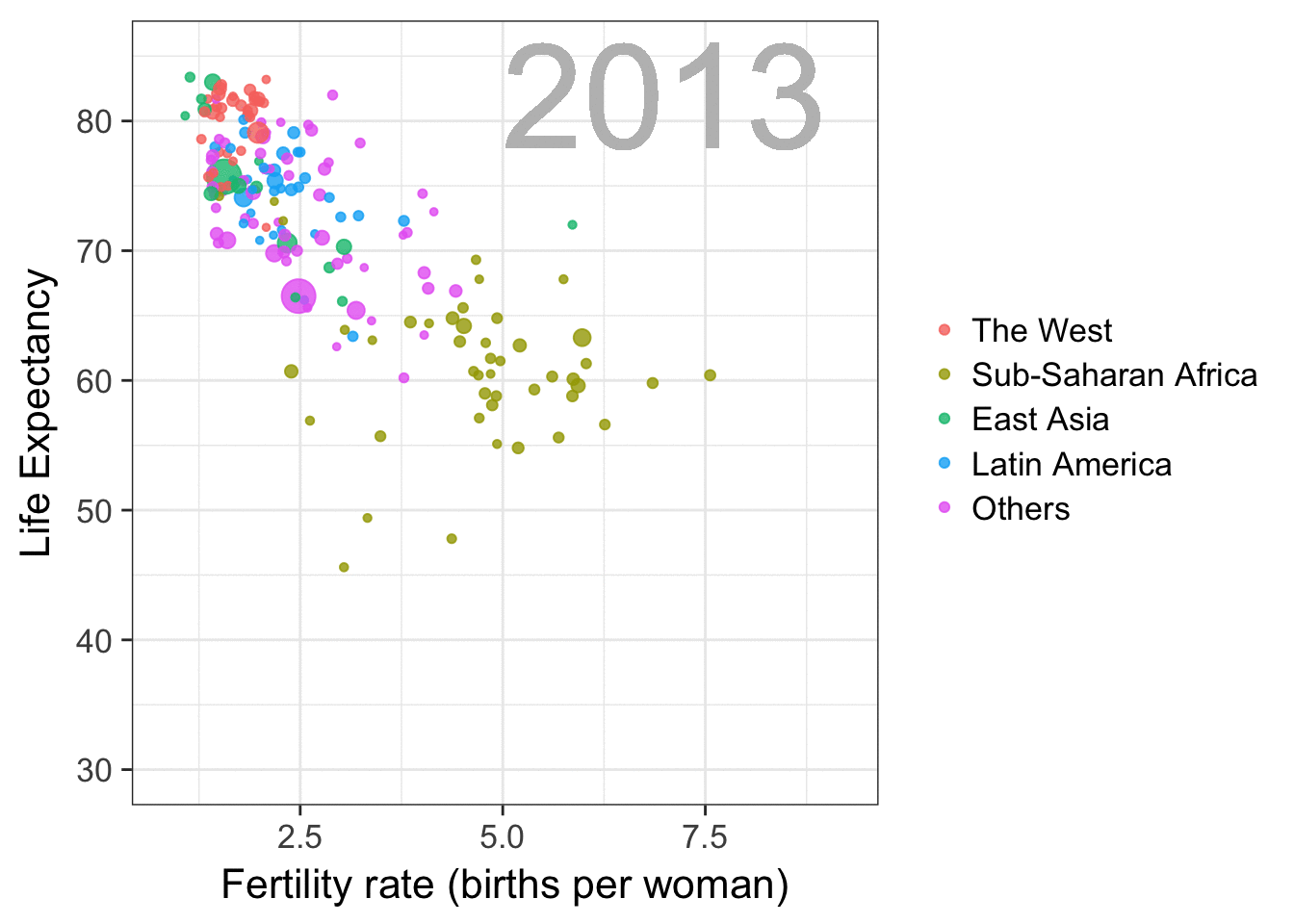
Comentatrios de las creadoras de flipbookr
flipbookr se actualiza para mejorar su funcionalidad
Comentatrios de las creadoras de flipbookr
flipbookr se actualiza para mejorar su funcionalidad
Puedes contribur en su construcción en el repositorioo de Eva: https://github.com/EvaMaeRey/flipbookr!
Crea tu flipbook
Crea tu flipbook
Prerequisitos
Crea tu flipbook
Prerequisitos
- Tener ganas de compartir datos y su "Transformación" de manera accesible.
Crea tu flipbook
Prerequisitos
Tener ganas de compartir datos y su "Transformación" de manera accesible.
Crea tu flipbook
Prerequisitos
Tener ganas de compartir datos y su "Transformación" de manera accesible.
Enseñar de manera incremental la creación de gráficas o cambio en los datos.
Crea tu flipbook
Prerequisitos
Tener ganas de compartir datos y su "Transformación" de manera accesible.
Enseñar de manera incremental la creación de gráficas o cambio en los datos.
Extra
Puedes hacer un solo chunk de código y a partir de una función se logra partir en secciones para presentar la evolución de un proceso.
Recomiendo que el chunk de código donde se encuentra lo que quieres presentar tenga en su encabezado el comando
include = F, es para que no se vea toda la información de código. Más detalles aquí
Ahora sí
Ahora sí
- Usa el comando
remotes::install_github("EvaMaeRey/flipbookr")
Ahora sí
Usa el comando
remotes::install_github("EvaMaeRey/flipbookr")Mandar a llamar la librería en el chunk inicial
library(flipbookr)
Ahora sí
Usa el comando
remotes::install_github("EvaMaeRey/flipbookr")Mandar a llamar la librería en el chunk inicial
library(flipbookr)Usar la función
chunk_reveal()para entender las distintas formas de dar a conocer el código de algún chunk.La función no se usa dentro del chunk, sino FUERA del chunk. Usa el formato 'r chunk_reveal()'
Ahora sí
Usa el comando
remotes::install_github("EvaMaeRey/flipbookr")Mandar a llamar la librería en el chunk inicial
library(flipbookr)Usar la función
chunk_reveal()para entender las distintas formas de dar a conocer el código de algún chunk.La función no se usa dentro del chunk, sino FUERA del chunk. Usa el formato 'r chunk_reveal()'
Dar formato de diseño con la función anterior.
Ahora sí
Usa el comando
remotes::install_github("EvaMaeRey/flipbookr")Mandar a llamar la librería en el chunk inicial
library(flipbookr)Usar la función
chunk_reveal()para entender las distintas formas de dar a conocer el código de algún chunk.La función no se usa dentro del chunk, sino FUERA del chunk. Usa el formato 'r chunk_reveal()'
Dar formato de diseño con la función anterior.
¡Disfruta el resultado!
Ejemplo. Pinguinos
Para demostrar cómo funciona flipbookr, usaremos datos sobre pinguinos.
Ejemplo a trabajar
Con el propósito de entender mejor la forma de crear un flipbook con R, usaré la información vertida sobre los pinguinos. Puedes aprender más de los datos.
#Instalar el paquete con los datosremotes::install_github("allisonhorst/palmerpenguins")#Mandar a llamar los datoslibrary(palmerpenguins)Ejemplo a trabajar
Con el propósito de entender mejor la forma de crear un flipbook con R, usaré la información vertida sobre los pinguinos. Puedes aprender más de los datos.
#Instalar el paquete con los datosremotes::install_github("allisonhorst/palmerpenguins")#Mandar a llamar los datoslibrary(palmerpenguins)Demos un vistazo a la información.
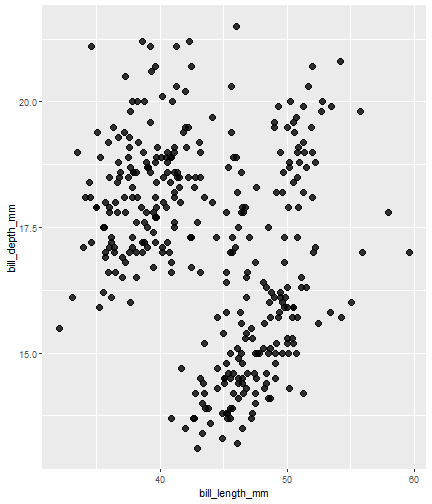
str(penguins)tibble [344 x 8] (S3: tbl_df/tbl/data.frame) $ species : Factor w/ 3 levels "Adelie","Chinstrap",..: 1 1 1 1 1 1 1 1 1 1 ... $ island : Factor w/ 3 levels "Biscoe","Dream",..: 3 3 3 3 3 3 3 3 3 3 ... $ bill_length_mm : num [1:344] 39.1 39.5 40.3 NA 36.7 39.3 38.9 39.2 34.1 42 ... $ bill_depth_mm : num [1:344] 18.7 17.4 18 NA 19.3 20.6 17.8 19.6 18.1 20.2 ... $ flipper_length_mm: int [1:344] 181 186 195 NA 193 190 181 195 193 190 ... $ body_mass_g : int [1:344] 3750 3800 3250 NA 3450 3650 3625 4675 3475 4250 ... $ sex : Factor w/ 2 levels "female","male": 2 1 1 NA 1 2 1 2 NA NA ... $ year : int [1:344] 2007 2007 2007 2007 2007 2007 2007 2007 2007 2007 ...head(penguins_raw)# A tibble: 6 x 17 studyName `Sample Number` Species Region Island Stage `Individual ID` <chr> <dbl> <chr> <chr> <chr> <chr> <chr> 1 PAL0708 1 Adelie Pengu~ Anvers Torger~ Adult,~ N1A1 2 PAL0708 2 Adelie Pengu~ Anvers Torger~ Adult,~ N1A2 3 PAL0708 3 Adelie Pengu~ Anvers Torger~ Adult,~ N2A1 4 PAL0708 4 Adelie Pengu~ Anvers Torger~ Adult,~ N2A2 5 PAL0708 5 Adelie Pengu~ Anvers Torger~ Adult,~ N3A1 6 PAL0708 6 Adelie Pengu~ Anvers Torger~ Adult,~ N3A2 # ... with 10 more variables: Clutch Completion <chr>, Date Egg <date>,# Culmen Length (mm) <dbl>, Culmen Depth (mm) <dbl>,# Flipper Length (mm) <dbl>, Body Mass (g) <dbl>, Sex <chr>,# Delta 15 N (o/oo) <dbl>, Delta 13 C (o/oo) <dbl>, Comments <chr>head(penguins_raw)# A tibble: 6 x 17 studyName `Sample Number` Species Region Island Stage `Individual ID` <chr> <dbl> <chr> <chr> <chr> <chr> <chr> 1 PAL0708 1 Adelie Pengu~ Anvers Torger~ Adult,~ N1A1 2 PAL0708 2 Adelie Pengu~ Anvers Torger~ Adult,~ N1A2 3 PAL0708 3 Adelie Pengu~ Anvers Torger~ Adult,~ N2A1 4 PAL0708 4 Adelie Pengu~ Anvers Torger~ Adult,~ N2A2 5 PAL0708 5 Adelie Pengu~ Anvers Torger~ Adult,~ N3A1 6 PAL0708 6 Adelie Pengu~ Anvers Torger~ Adult,~ N3A2 # ... with 10 more variables: Clutch Completion <chr>, Date Egg <date>,# Culmen Length (mm) <dbl>, Culmen Depth (mm) <dbl>,# Flipper Length (mm) <dbl>, Body Mass (g) <dbl>, Sex <chr>,# Delta 15 N (o/oo) <dbl>, Delta 13 C (o/oo) <dbl>, Comments <chr>Ambos conjuntos de datos contienen datos de 344 pingüinos, sin smbargo el primero tiene menos variables que el segundo.
Hay 3 especies diferentes de pingüinos en este conjunto de datos, recopilados en 3 islas del archipiélago de Palmer, en la Antártida.

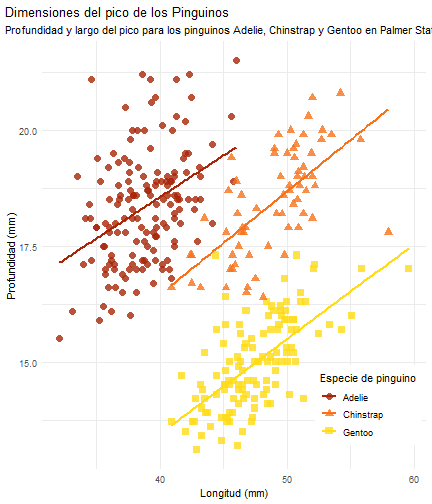

Con este gráfico y otros estaremos trabajando los flipbooks.
Flipbook muestra
penguins# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>Flipbook muestra
penguins %>% ggplot()
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm)
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)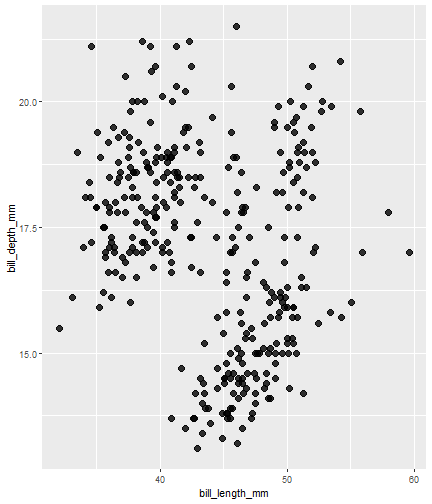
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)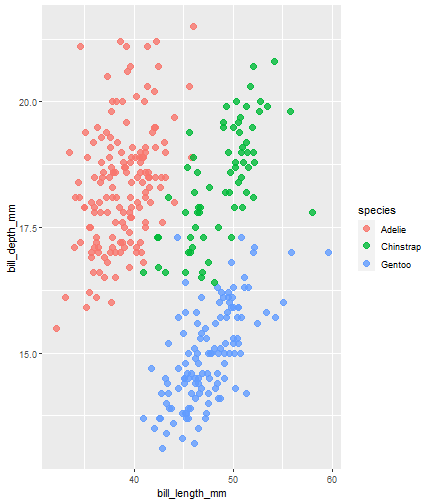
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)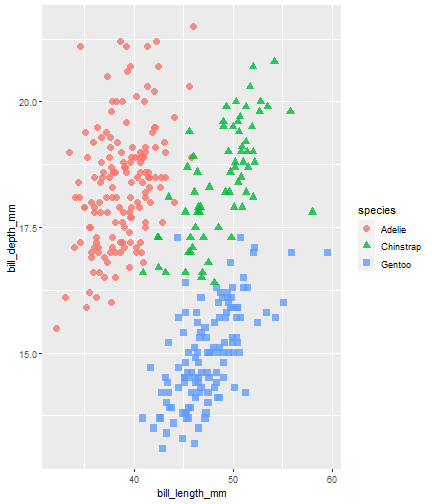
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)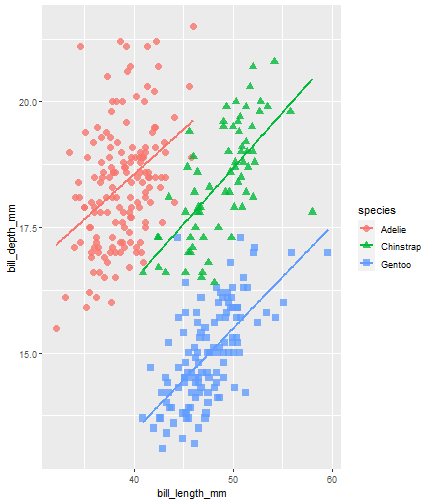
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)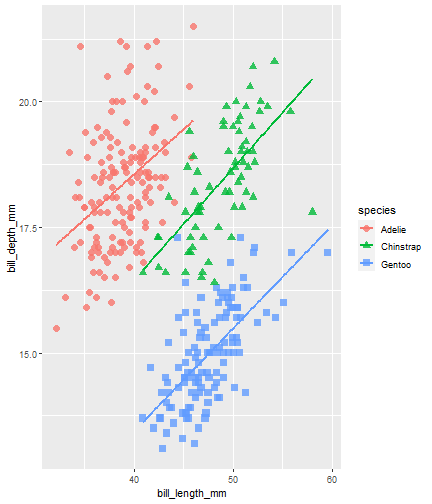
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()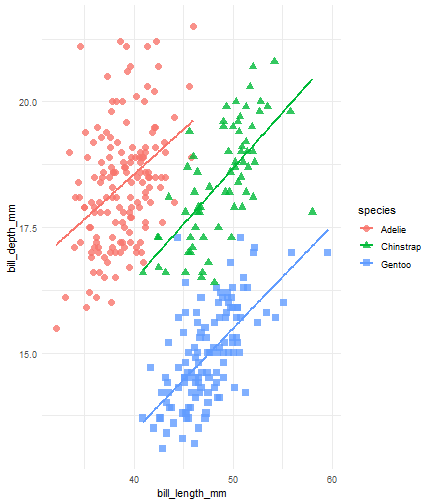
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))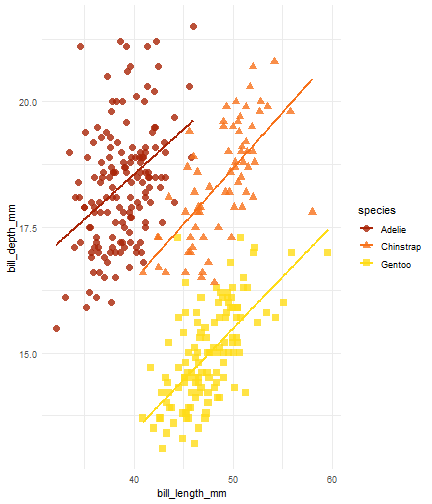
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")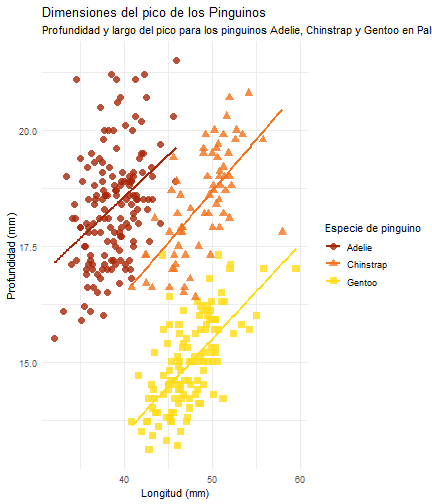
Flipbook muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")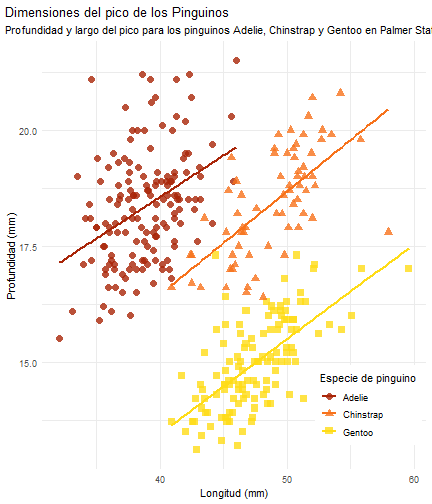
La función importante chunk_reveal()
La función chunk_reveal() sirve para hacer los chunks de código legibles por partes de tal forma que el entendimiento del código sea correcto. En la presentación de Xaringan se vería:
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Existen muchas formas de mostrar en modo "flipbook" tu código, lo cual veremos a continuación con los...
Parámetros
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Parámetros
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Los siguientes parámetros son fundamentales para entender la función:
Parámetros
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Los siguientes parámetros son fundamentales para entender la función:
- chunk_name se le asigna el nombre del chunk de código, del ejemplo "cars". Para asignar el nombre a un chunk de código, solo es necesario escribirlo al lado de la letra r:
Parámetros
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Los siguientes parámetros son fundamentales para entender la función:
- chunk_name se le asigna el nombre del chunk de código, del ejemplo "cars". Para asignar el nombre a un chunk de código, solo es necesario escribirlo al lado de la letra r:

- break_type indica qué linea del código del chunk se revelará y en qué momento. Por defecto el parámetro tiene la asignación "auto". Es decir, sino especificas su valor, entonces el valor por defecto será "auto".
Parámetros
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Los siguientes parámetros son fundamentales para entender la función:
- chunk_name se le asigna el nombre del chunk de código, del ejemplo "cars". Para asignar el nombre a un chunk de código, solo es necesario escribirlo al lado de la letra r:

break_type indica qué linea del código del chunk se revelará y en qué momento. Por defecto el parámetro tiene la asignación "auto". Es decir, sino especificas su valor, entonces el valor por defecto será "auto".
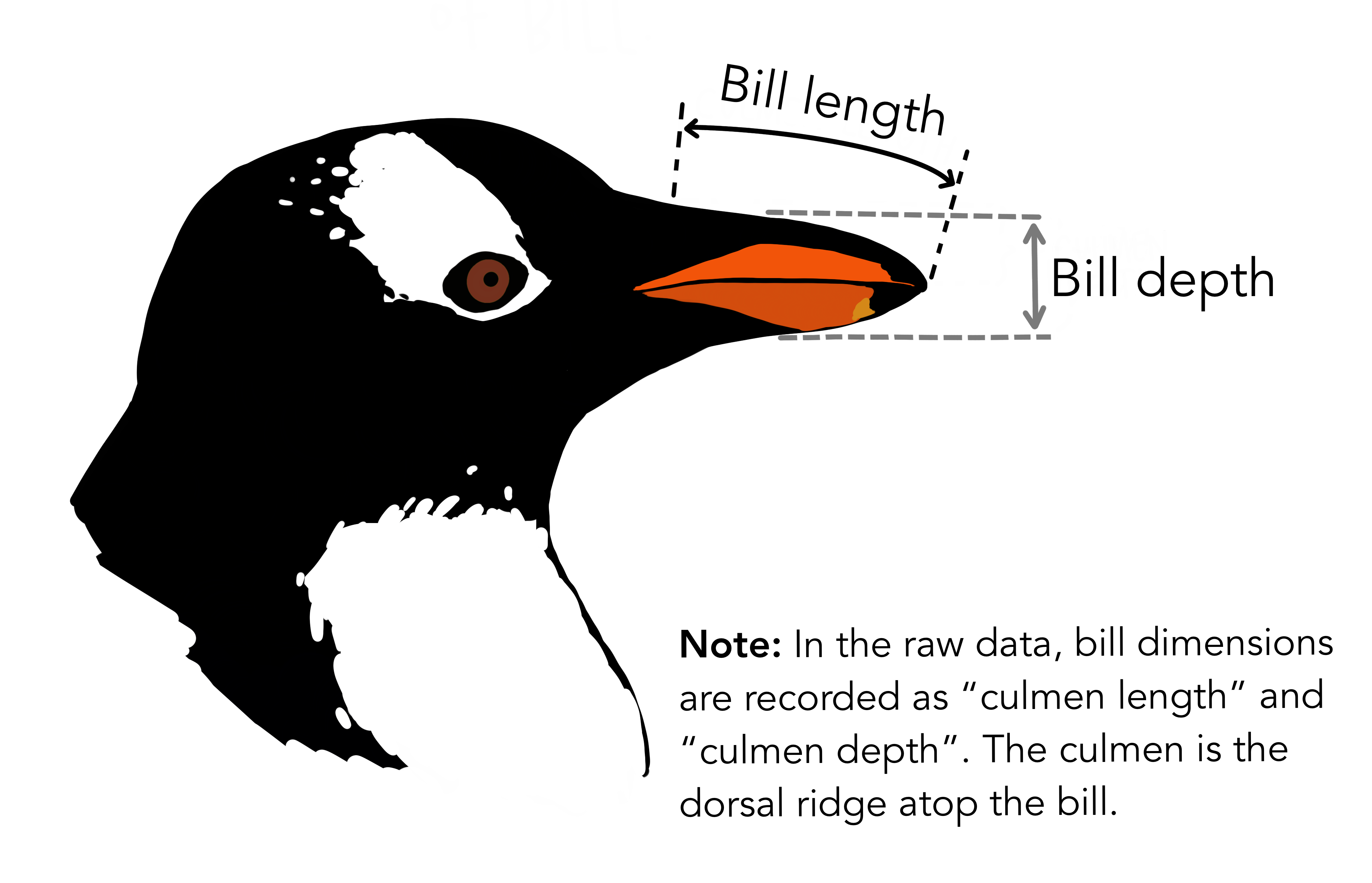
display_type indica si se desea que "se vea" en la presentación el codigo, el resultado del código o ambos. Por defecto
display_type="both".
Parámetros
`r chunk_reveal(chunk_name = "cars", break_type = "user")`Los siguientes parámetros son fundamentales para entender la función:
- chunk_name se le asigna el nombre del chunk de código, del ejemplo "cars". Para asignar el nombre a un chunk de código, solo es necesario escribirlo al lado de la letra r:

break_type indica qué linea del código del chunk se revelará y en qué momento. Por defecto el parámetro tiene la asignación "auto". Es decir, sino especificas su valor, entonces el valor por defecto será "auto".
display_type indica si se desea que "se vea" en la presentación el codigo, el resultado del código o ambos. Por defecto
display_type="both".left_assign Cuando left_assign = TRUE, el primer objeto creado se imprime al final del chunk de código.
Veremos en esta presentación las diferentes formas de usar estos parámetros así como sus resultados.
break_type
break_type
Este parámetro de la función chunk_reveal() ayuda a distinguir cuándo aparece el código o resultado del que queremos hacer un flipbook
break_type
Las opciones de este parámetro son:
auto
user
non_seq
rotate
n (algún número natural)
Recordemos que este parámetro permite que quien crea el flipbook decida cuál parte del código desea que aparezca primero, depués, etc.
break_type
Las opciones de este parámetro son:
auto
user
non_seq
rotate
n (algún número natural)
Recordemos que este parámetro permite que quien crea el flipbook decida cuál parte del código desea que aparezca primero, depués, etc.
Veremos con los datos de los pinguinos cómo funcionan estas opciones.
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAKbreak_type = "auto"
El valor "auto" del parámetro break_type se da por defecto sino se especifica otra opción. La forma en que chunk_reveal() decide cómo mostrar las partes del código es por orden de aparición de los paréntesis o pipes (%>%).
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAKbreak_type = auto
penguins# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>break_type = auto
penguins %>% ggplot()
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")
break_type = auto
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")
break_type = "user", con #BREAK
Tú puedes decidir cómo quieres que aparezcan las partes del código, esto se hace con la opción "user" y #BREAK.
En donde aparezca #BREAK se detendrá la aparición del código (se observan los resultados del código hasta el BREAK). Si hay más de un #BREAK, entonces el código aparecerá en ese orden.
break_type = "user", con #BREAK
Tú puedes decidir cómo quieres que aparezcan las partes del código, esto se hace con la opción "user" y #BREAK.
En donde aparezca #BREAK se detendrá la aparición del código (se observan los resultados del código hasta el BREAK). Si hay más de un #BREAK, entonces el código aparecerá en ese orden.
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAKbreak_type = user
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)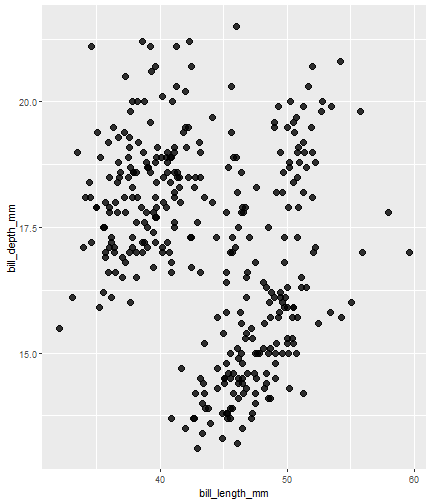
break_type = user
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()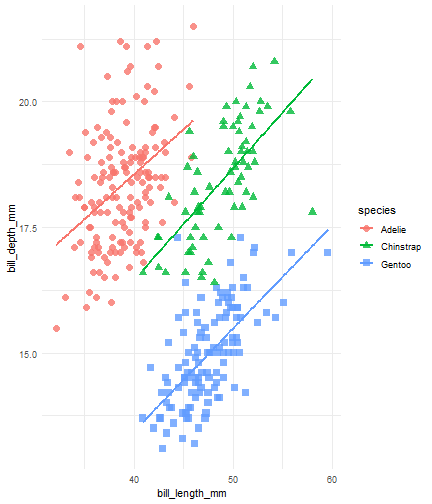
break_type = user
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")
break_type = user
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")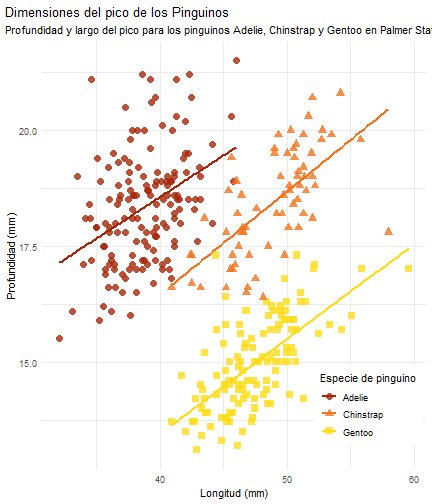
break_type = "non_seq", con #BREAK2, #BREAK3
Es parecido a la opción anterior pero es necesario el uso de números después del #BREAK.
El número indica el momento en que aparecerá y se ejecutará esa línea de código.
Primero aparecerá todo lo que no tenga #BREAK N y luego aparecerá la línea donde N = 2, luego N = 3 y así en lo sucesivo.
break_type = "non_seq", con #BREAK2, #BREAK3
Es parecido a la opción anterior pero es necesario el uso de números después del #BREAK.
El número indica el momento en que aparecerá y se ejecutará esa línea de código.
Primero aparecerá todo lo que no tenga #BREAK N y luego aparecerá la línea donde N = 2, luego N = 3 y así en lo sucesivo.
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAKbreak_type = non_seq
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")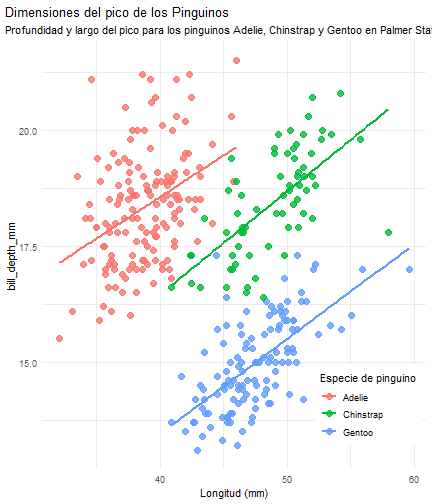
break_type = non_seq
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")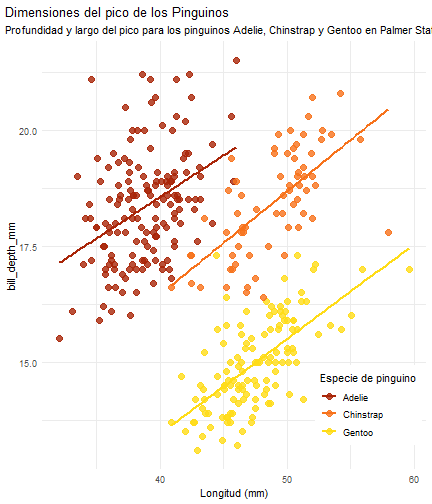
break_type = non_seq
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")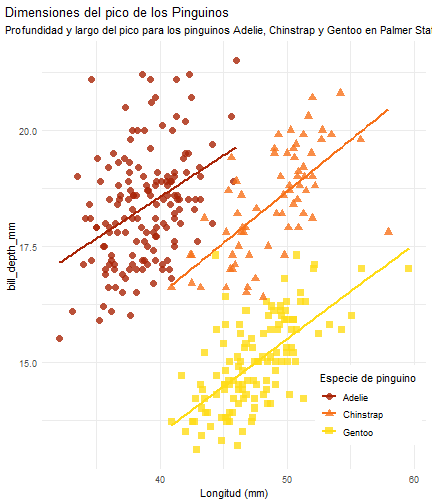
break_type = non_seq
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")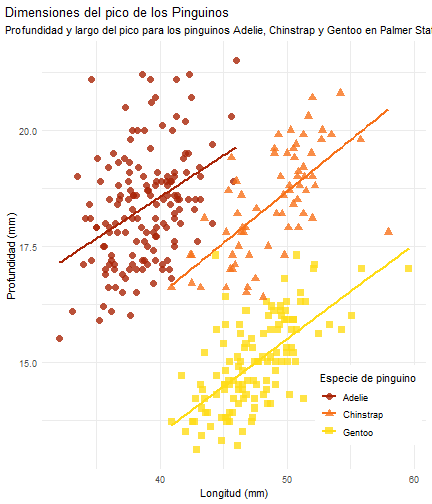
break_type = "rotate"
Si queremos dar "varias opciones" a la audiencia sobre las formas en que pueden tratar su código, usamos este tipo de aparición con la palabra #ROTATE posterior a la línea de código que deseamos de esa forma presentar.
Dado que en el chunk de código aparecerán las opciones de la misma idea de codficación, éste no se desea evaluar pues aparecería un error. Para solucionar esto, usamos echo = FALSE.
break_type = "rotate"
Si queremos dar "varias opciones" a la audiencia sobre las formas en que pueden tratar su código, usamos este tipo de aparición con la palabra #ROTATE posterior a la línea de código que deseamos de esa forma presentar.
Dado que en el chunk de código aparecerán las opciones de la misma idea de codficación, éste no se desea evaluar pues aparecería un error. Para solucionar esto, usamos echo = FALSE.
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal() + labs(x = "Flipper length (mm)", y = "Frequency", title = "Penguin flipper lengths") + scale_fill_manual(values = c("darkorange","purple","cyan4")) + #ROTATE scale_fill_manual(values = c("#09A901","#337D2F","#67F11D")) + #ROTATE scale_fill_manual(values = c("#F11D94","#6A3401","#F3F185")) #ROTATEbreak_type = rotate
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal() + labs(x = "Flipper length (mm)", y = "Frequency", title = "Penguin flipper lengths") + scale_fill_manual(values = c("darkorange","purple","cyan4"))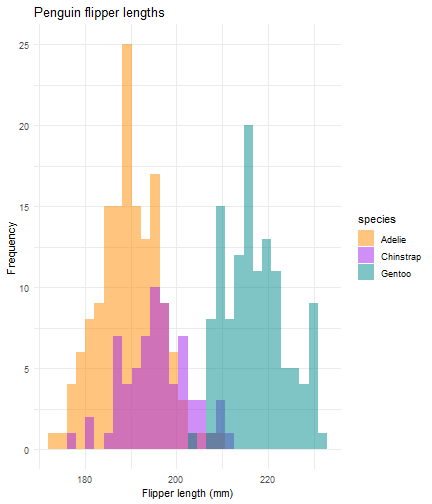
break_type = rotate
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal() + labs(x = "Flipper length (mm)", y = "Frequency", title = "Penguin flipper lengths") + scale_fill_manual(values = c("#09A901","#337D2F","#67F11D"))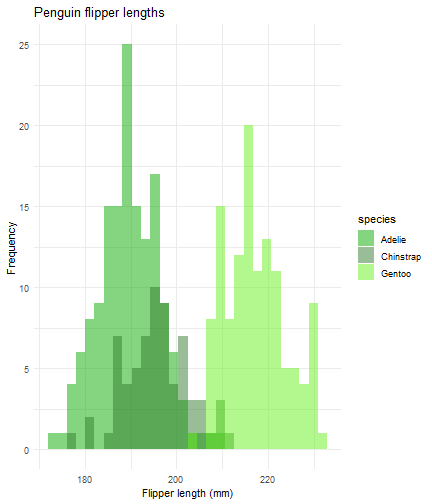
break_type = rotate
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal() + labs(x = "Flipper length (mm)", y = "Frequency", title = "Penguin flipper lengths") + scale_fill_manual(values = c("#F11D94","#6A3401","#F3F185"))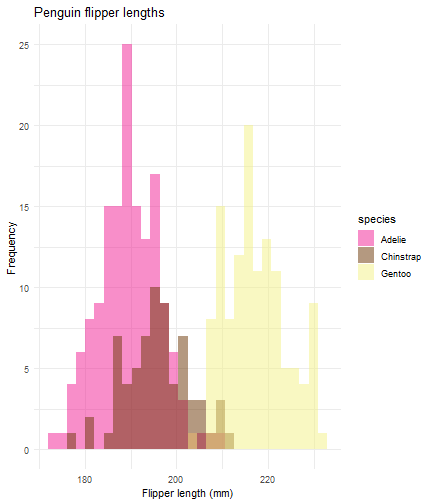
Posibles combinaciones
Si deseas que aparezca el código paulatinamente y además se cambie una sección particular del código, solo es necesario añadir el parámetro omit dentro de la función chunk_reveal() con el valor "#ROTATE".
Escribimos r chunk_reveal("omit", omit = "#ROTATE", break_type = "auto") en la "diapositiva" donde queremos que aparezca la combinación.
En la diapositiva posterior escribimos r chunk_reveal("omit", omit = "#ROTATE", break_type = "auto") para que se de la "rotación".
Posibles combinaciones
Si deseas que aparezca el código paulatinamente y además se cambie una sección particular del código, solo es necesario añadir el parámetro omit dentro de la función chunk_reveal() con el valor "#ROTATE".
Escribimos r chunk_reveal("omit", omit = "#ROTATE", break_type = "auto") en la "diapositiva" donde queremos que aparezca la combinación.
En la diapositiva posterior escribimos r chunk_reveal("omit", omit = "#ROTATE", break_type = "auto") para que se de la "rotación".
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#FF7400","#944300","#341800")) + #OMIT scale_fill_manual(values = c("darkorange","purple","cyan4")) + #ROTATE scale_fill_manual(values = c("#09A901","#337D2F","#67F11D")) + #ROTATE scale_fill_manual(values = c("#F11D94","#6A3401","#F3F185")) + #ROTATE theme_minimal() + labs(x = "Flipper length (mm)") + labs(y = "Frequency") + labs(title = "Penguin flipper lengths")Combinaciones rotate-incremental
ggplot(data = penguins)
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm)
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity")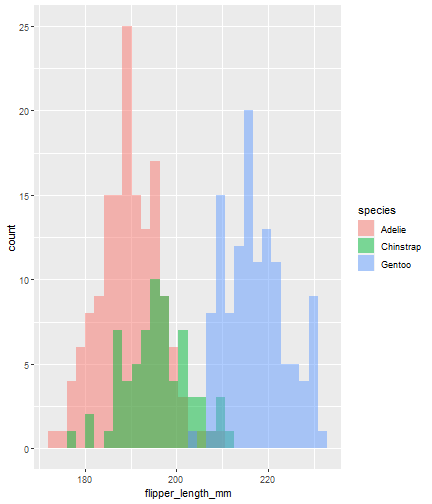
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#FF7400","#944300","#341800"))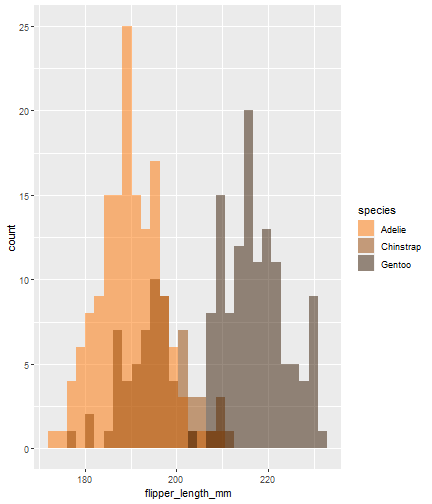
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#FF7400","#944300","#341800")) + theme_minimal()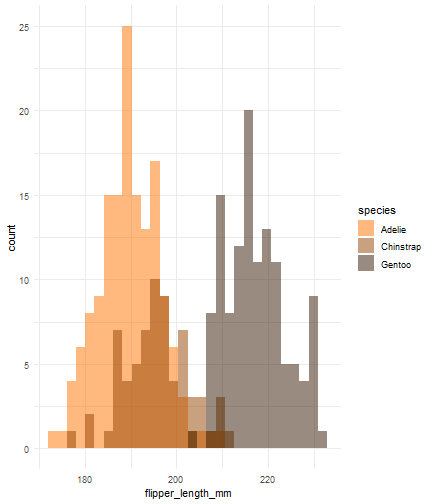
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#FF7400","#944300","#341800")) + theme_minimal() + labs(x = "Flipper length (mm)")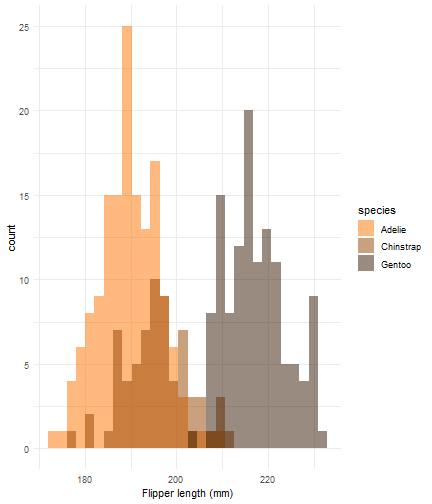
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#FF7400","#944300","#341800")) + theme_minimal() + labs(x = "Flipper length (mm)") + labs(y = "Frequency")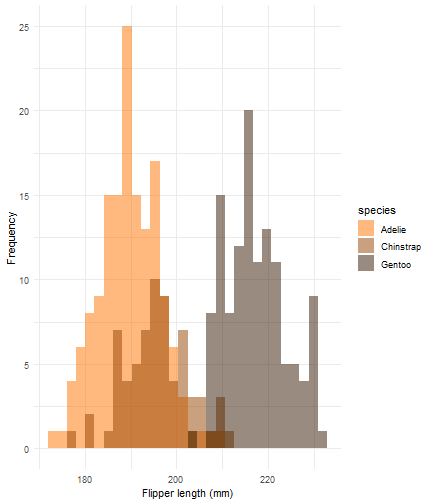
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#FF7400","#944300","#341800")) + theme_minimal() + labs(x = "Flipper length (mm)") + labs(y = "Frequency") + labs(title = "Penguin flipper lengths")
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("darkorange","purple","cyan4")) + theme_minimal() + labs(x = "Flipper length (mm)") + labs(y = "Frequency") + labs(title = "Penguin flipper lengths")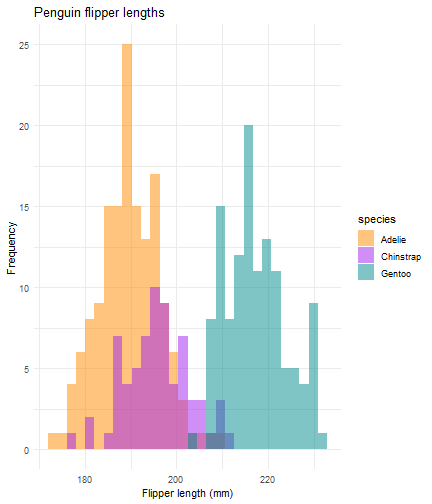
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#09A901","#337D2F","#67F11D")) + theme_minimal() + labs(x = "Flipper length (mm)") + labs(y = "Frequency") + labs(title = "Penguin flipper lengths")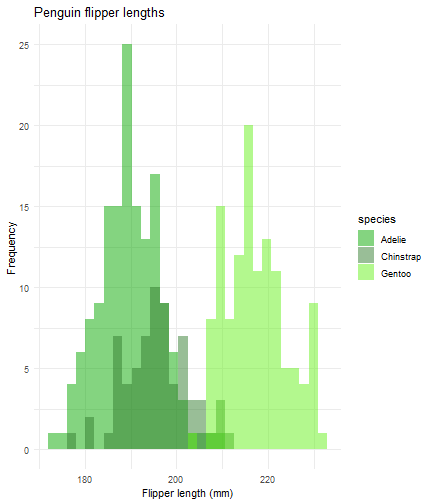
Combinaciones rotate-incremental
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + scale_fill_manual(values = c("#F11D94","#6A3401","#F3F185")) + theme_minimal() + labs(x = "Flipper length (mm)") + labs(y = "Frequency") + labs(title = "Penguin flipper lengths")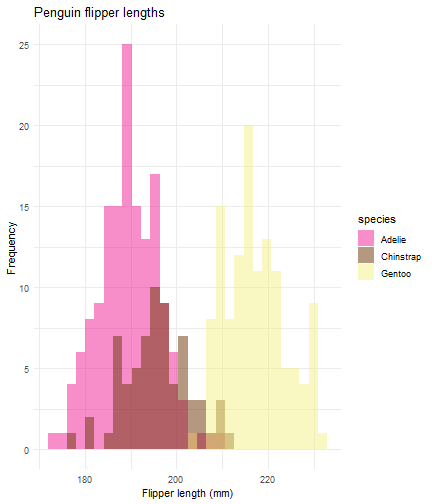
break_type = n
Si tenemos una muestra aleatoria de la cual queremos hacer un mismo análisis entonces es conveniente usar break_type = n. Esto nos permitirá enseñar la forma en que cambia el gráfico asociado al código con cada muestra diferente hasta n clicks.
Si n = 1 entonces tendremos el código simple y su resultado.
break_type = n
Si tenemos una muestra aleatoria de la cual queremos hacer un mismo análisis entonces es conveniente usar break_type = n. Esto nos permitirá enseñar la forma en que cambia el gráfico asociado al código con cada muestra diferente hasta n clicks.
Si n = 1 entonces tendremos el código simple y su resultado.
proceso_markov_TPH <- function(){ #Trayectoria X X <- NULL #Pasos a simular: M #El tiempo de permanencia en cada estado (por cada salto) Tn <- c() #El estado donde inicia la trayectoria X[1] <- sample(c(1, 2, 3, 4, 5, 6), 1, replace = TRUE, prob = vec_pi) Tn[1] <- rexp(1, lambdas[X[1]]) i=1 while(T) { i <- i+1 #Dado que tenemos un estado inicial X[1], elegimos otro estado #a partir de la matriz de probabilidades de transición Pij X[i] <- sample(c(1, 2, 3, 4, 5, 6), 1, replace = T , prob = P[X[i - 1], ]) #Una vez definido el estado al que "saltará" la cadena, definimos el #tiempo de permanencia en el mismo con las "lambdas" correspondientes if (X[i] == 1) { Tn[i] <- rexp(1, lambdas[1]) } else if (X[i] == 2) { Tn[i] <- rexp(1, lambdas[2]) } else if (X[i] == 3) { Tn[i] <- rexp(1, lambdas[3]) } else if (X[i] == 4) { Tn[i] <- rexp(1, lambdas[4]) } else if (X[i] == 5) { Tn[i] <- rexp(1, lambdas[5]) } else{ break } } #Creamos la trayectoria que inicie en cero (en cuando al tiempo) y posteriormente haga los #"saltos" en cada tiempo acumulado de los saltos que van hasta el momento. trayectoria <- data.frame( x = c(0, cumsum(Tn)), y = X, xend = c(cumsum(Tn), sum(Tn) + .5), yend = X ) grafica <- ggplot(trayectoria, aes( x = x, y = y, xend = xend, yend = yend )) + geom_segment(color = 'orange', lwd = 1.3) + ggtitle("Trayectoria de la cadena de Markov asociada a una v.a. Tipo Fase") + geom_point(color = 'red') + xlab("Tiempo") + ylab("Estados") + theme( panel.background = element_rect(fill = "white"), strip.background = element_rect(fill = "black") ) grafica <- grafica + geom_segment( x = sum(Tn), xend = (sum(Tn) + .5), y = 6, yend = 6, color = "violet", lwd = 1.4 )+geom_point(x = sum(Tn),y = 6, color = 'purple') return(list(grafica, sum(Tn)))}break_type = 7
proceso_markov_TPH()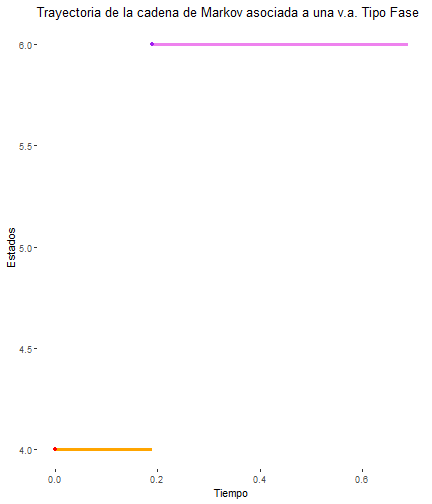
break_type = 7
proceso_markov_TPH()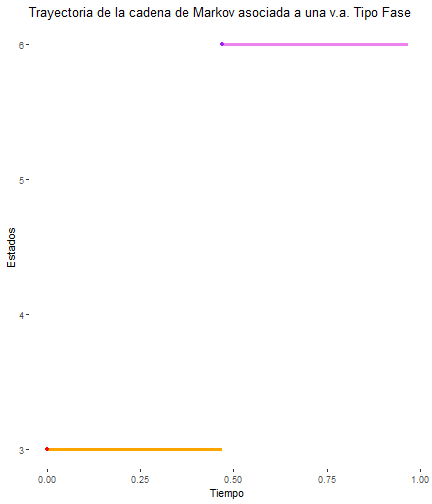
break_type = 7
proceso_markov_TPH()
break_type = 7
proceso_markov_TPH()
break_type = 7
proceso_markov_TPH()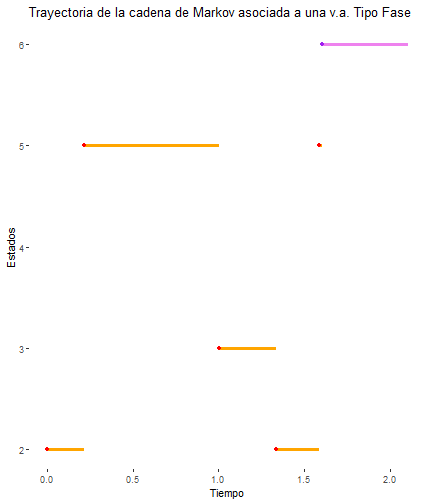
break_type = 7
proceso_markov_TPH()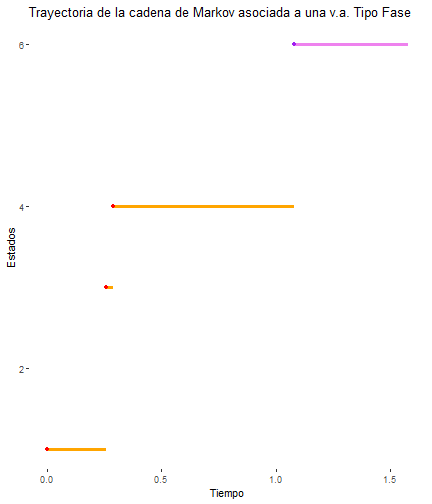
break_type = 7
proceso_markov_TPH()
proceso_markov_TPH()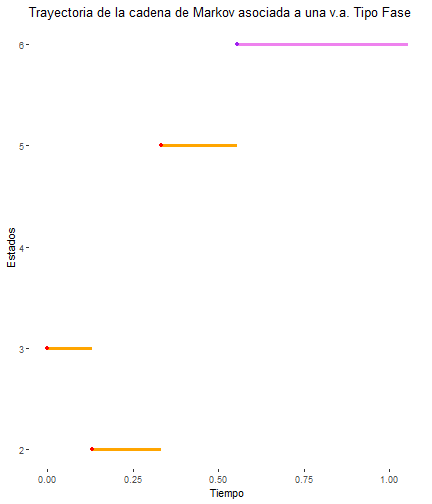
break_type = "replace"
Si queremos que los valores dentro del chunk de código sean remplazados varias veces para mostrar algo, entonces esta opción del break_type puede interesar.
Añadir el argumento
replace = "lo_que_quieres_remplazar"Poner además
replacements = lo_que_irá_en_vez_de_lo_que_quieres_remplazar
Nota: Puedes hacer tantos remplazos como quieras. Al i-ésimo remplazo le pondremos replacei = "lo_que_quieres_remplazar_por_i-ésima_vez" donde i>1.
break_type = "replace"
Si queremos que los valores dentro del chunk de código sean remplazados varias veces para mostrar algo, entonces esta opción del break_type puede interesar.
Añadir el argumento
replace = "lo_que_quieres_remplazar"Poner además
replacements = lo_que_irá_en_vez_de_lo_que_quieres_remplazar
Nota: Puedes hacer tantos remplazos como quieras. Al i-ésimo remplazo le pondremos replacei = "lo_que_quieres_remplazar_por_i-ésima_vez" donde i>1.
break_type = replace
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("cadetblue", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAK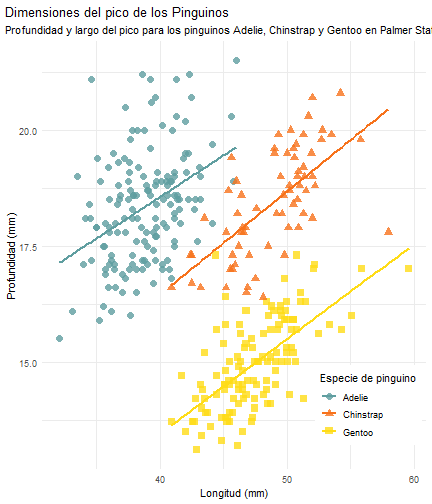
break_type = replace
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("darkolivegreen", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAK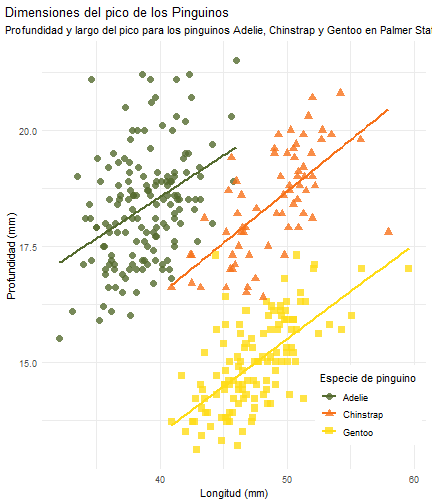
break_type = replace
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("firebrick2", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAK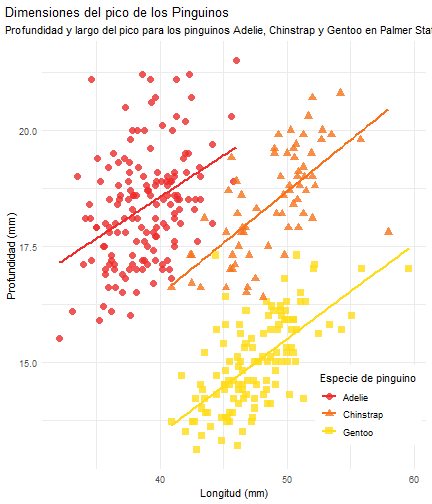
break_type = replace
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + #BREAK aes(color = species) + aes(shape = species) + #BREAK3 geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + #BREAK scale_color_manual(values = c("firebrick", "#F7721B", "#FFDC17")) + #BREAK2 labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", #BREAK4 color = "Especie de pinguino", shape = "Especie de pinguino") + #BREAK theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot") #BREAK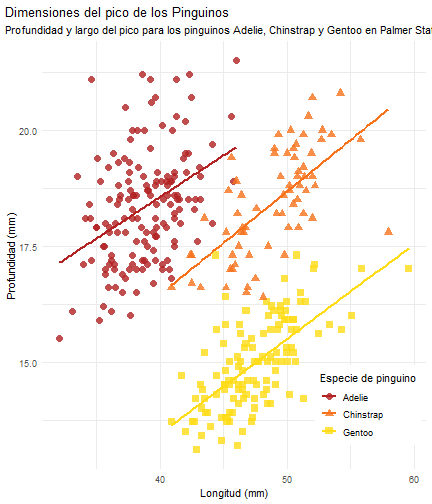
display_type
display_type
Este parámetro de la función chunk_reveal() ayuda a distinguir qué queremos que aparezca del flipbook. (Código, resultados, explicaciones,...)
display_type
Otro de los parámetros de la función chunk_reveal() es display_type al cual se le pueden asignar también diversas opciones:
Puede ser un vector de a lo más 3 entradas que sean alguna(s) de las siguientes:
- "code"
- "output"
- "func"
- "output_lag"
- "code_lag"
display_type
Otro de los parámetros de la función chunk_reveal() es display_type al cual se le pueden asignar también diversas opciones:
Puede ser un vector de a lo más 3 entradas que sean alguna(s) de las siguientes:
- "code"
- "output"
- "func"
- "output_lag"
- "code_lag"
Por defecto display_type = c("code", "output") que permite la forma normal del flipbook, es decir, el código y su resultado de manera progresiva y simultánea.
Además de las opciones anteriores, "md" también se puede usar pero requiere que le agregues otro parámetro a la función chunk_reveal() que se llama md y sirve para que selecciones lo que aparecerá en cada click en la presentación.
display_type
Otro de los parámetros de la función chunk_reveal() es display_type al cual se le pueden asignar también diversas opciones:
Puede ser un vector de a lo más 3 entradas que sean alguna(s) de las siguientes:
- "code"
- "output"
- "func"
- "output_lag"
- "code_lag"
Por defecto display_type = c("code", "output") que permite la forma normal del flipbook, es decir, el código y su resultado de manera progresiva y simultánea.
Además de las opciones anteriores, "md" también se puede usar pero requiere que le agregues otro parámetro a la función chunk_reveal() que se llama md y sirve para que selecciones lo que aparecerá en cada click en la presentación.
¡Veamos las opciones y sus resultados (outputs)!
display_type = "output"
Al usar esta opción como valor del parámetro display_type, solo se mostrará el resultado del código chunk de manera progresiva.
El break_type usado es el que está por defecto, es decir, "auto"
Sólo el resultado se muestra
# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

Sólo el resultado se muestra

display_type = "code"
Análogo a la opción "output", la opción "code" permitirá que solo se visualize la transición del código.
Sólo el código se muestra
penguinsSólo el código se muestra
penguins %>% ggplot()Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")Sólo el código se muestra
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")display_type = c("output", "code")
Si queremos cambiar la posición entre código y su resultado, simplemente intercambiamos las entradas de los vectores.
display_type = c("output", "code")
Si queremos cambiar la posición entre código y su resultado, simplemente intercambiamos las entradas de los vectores.
Recoremos que por defecto, la entrada del parámetro display_type permite que del lado izquierdo se vea el código y del derecho el resultado que se da a partir de ese código.
Invertimos el orden de la salida del flipbook
# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>penguinsInvertimos el orden de la salida del flipbook

penguins %>% ggplot()Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")Invertimos el orden de la salida del flipbook

penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")display_type = c("code", "output", "output_lag")
Agreguemos otra entrada al vector
Con esta opción podemos lograr que:
- Se vea el código.
- Se vea la salida que el código está ejecutando.
- Se vea la salida previa de ejecución del código.
Es decir, que aparecerán tres columnas de resultados.
La utilidad de esta opción es de la comparación que se puede dar de los resultados.
display_type = c("code", "output", "output_lag")
Agreguemos otra entrada al vector
Con esta opción podemos lograr que:
- Se vea el código.
- Se vea la salida que el código está ejecutando.
- Se vea la salida previa de ejecución del código.
Es decir, que aparecerán tres columnas de resultados.
La utilidad de esta opción es de la comparación que se puede dar de los resultados.
Como tenemos tres columnas de resultados, queremos que aparezcan con cierto ancho cada una, lo cual se hace con el parámetro widths el cual es un vector de tres entradas cada una de las cuales es un número que representa el ancho de las columnas.
Retraso de un resultado
penguins# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>[1] "--"Retraso de un resultado
penguins %>% ggplot()
# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm)

Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)

Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)

Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)

Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)
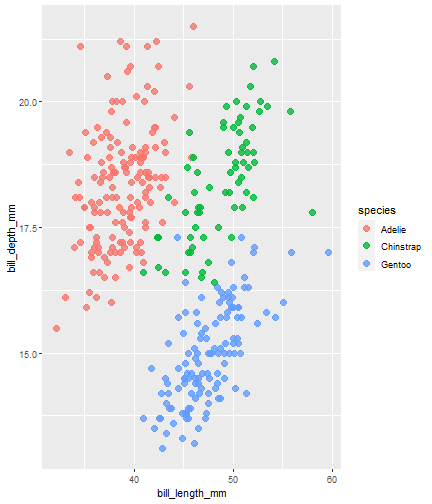
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)
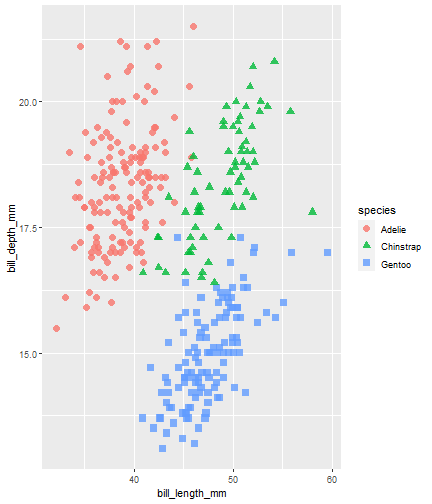
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)
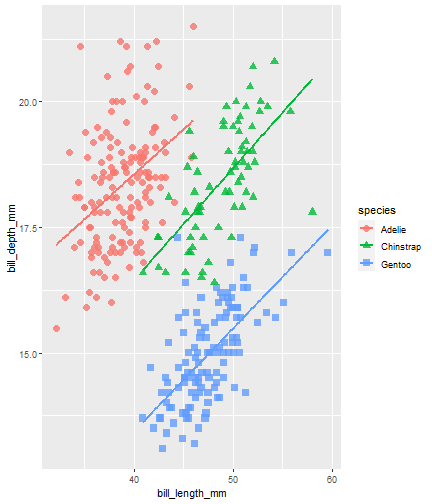
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()
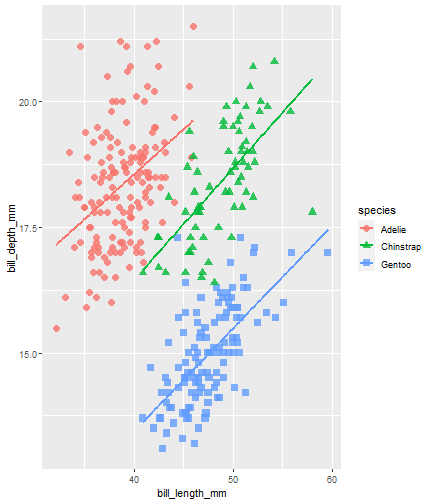
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))
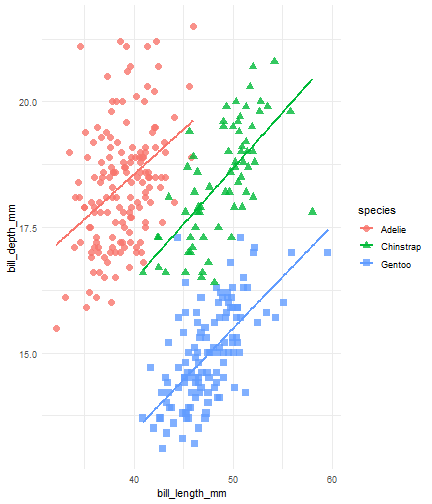
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")
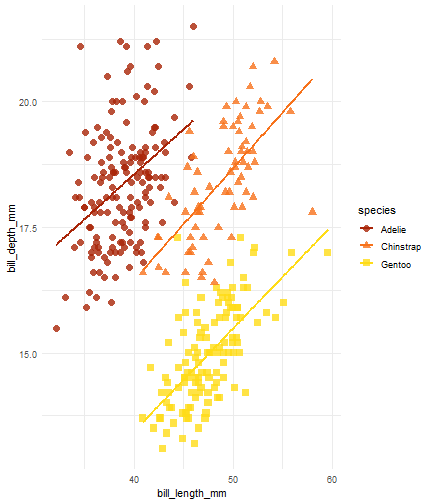
Retraso de un resultado
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")
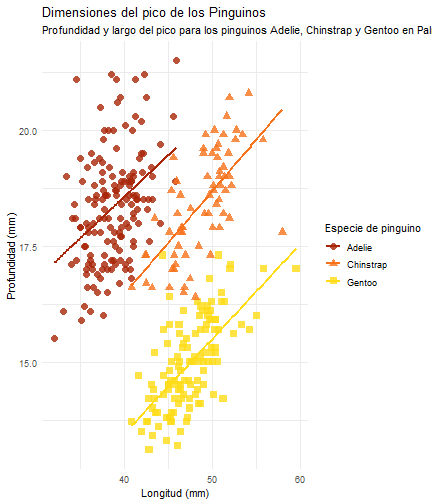
display_type = c("code", "output", "output_target")
Posiblemente deseamos que aparezca en cada momento el resultado al que aspiramos llegar. Esto se obtiene con "output_target".
display_type = c("code", "output", "output_target")
Posiblemente deseamos que aparezca en cada momento el resultado al que aspiramos llegar. Esto se obtiene con "output_target".
La ventaja del uso de esta entrada del vector asignado a display_type es que permites hacer un spoiler del resultado a la vez que muestras la forma en que se va construyendo el mismo paso a paso.
Ver a lo que queremos llegar
penguins# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>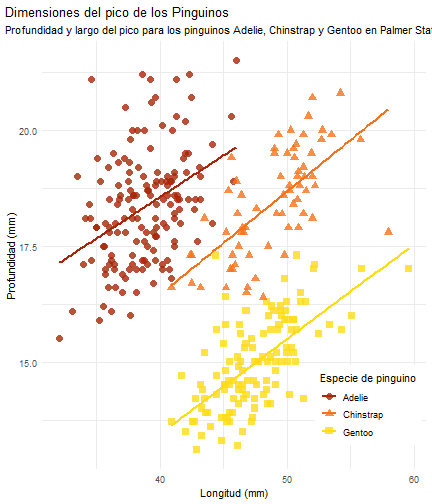
Ver a lo que queremos llegar
penguins %>% ggplot()
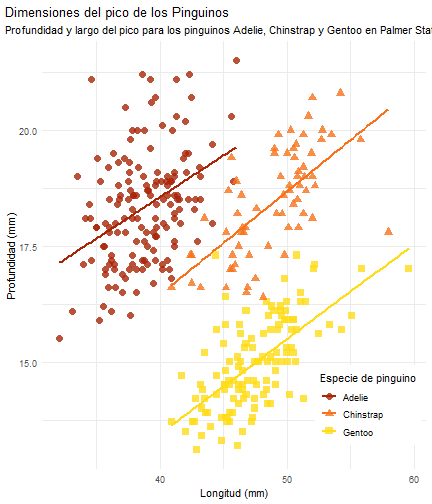
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm)
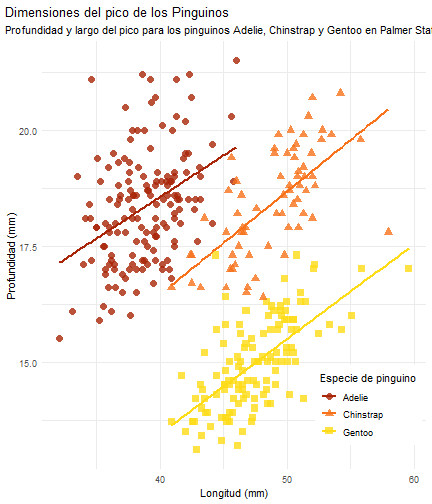
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)
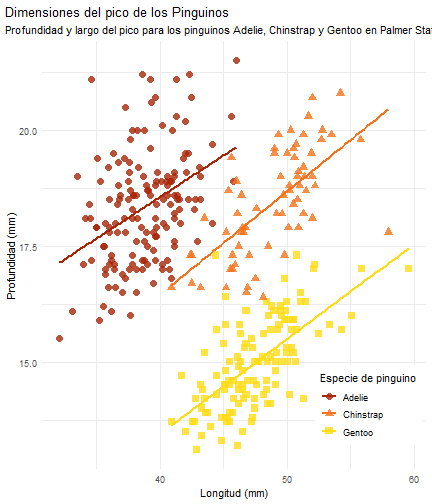
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)
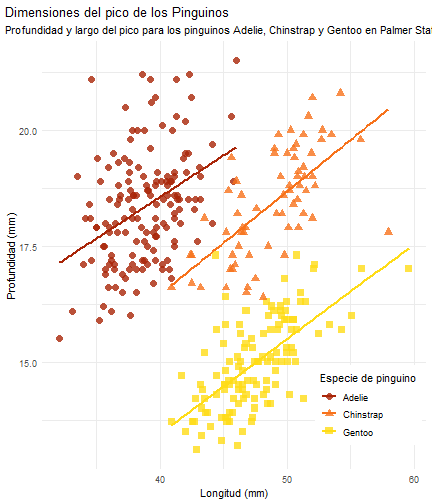
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)
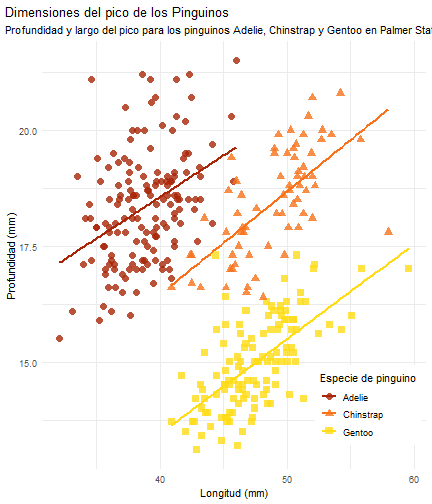
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)
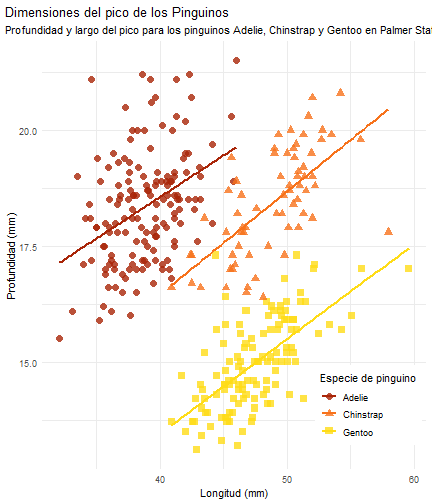
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)
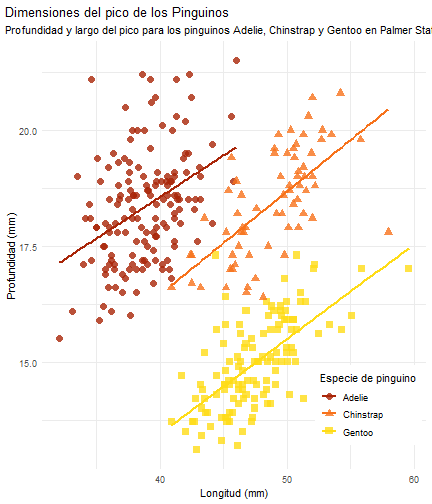
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)
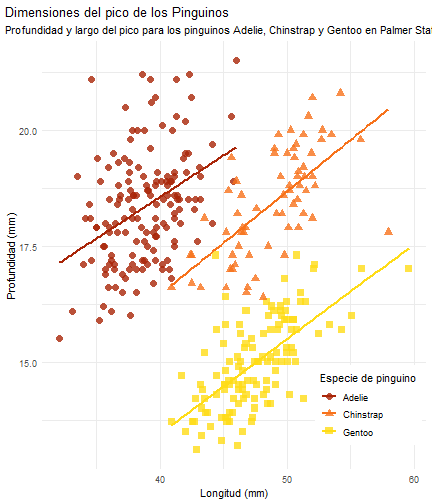
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)
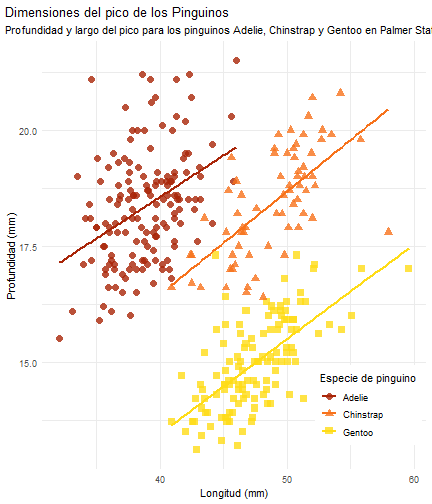
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()
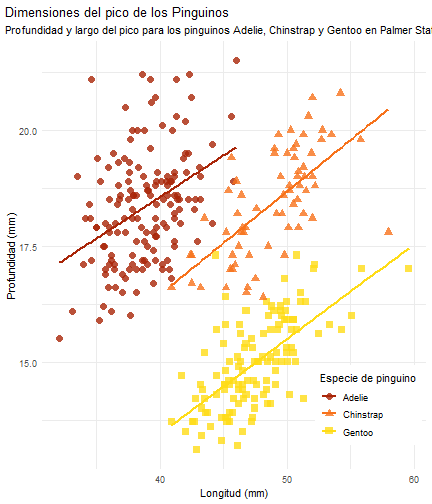
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))
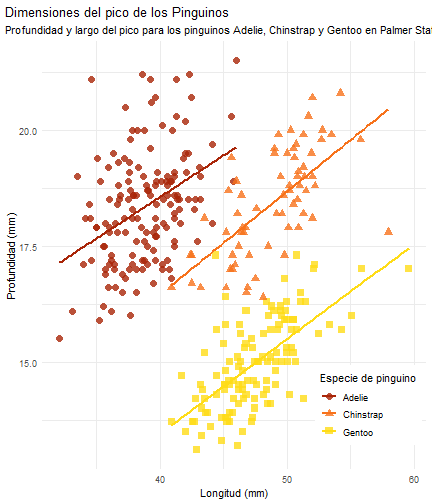
Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")

Ver a lo que queremos llegar
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")
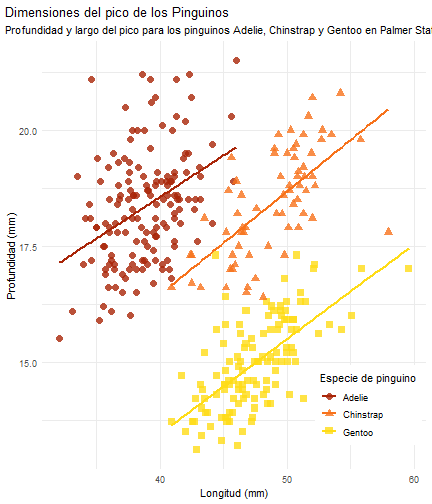
display_type = c("code", "output","output_start")
La diferencia entre esta salida y las anteriores es la entrada tres del vector asignado al parámetro display_type.
Cuando se usa "output_start", queremos que ahora aparezca en todo momento el resultado inicial del código.
display_type = c("code", "output","output_start")
La diferencia entre esta salida y las anteriores es la entrada tres del vector asignado al parámetro display_type.
Cuando se usa "output_start", queremos que ahora aparezca en todo momento el resultado inicial del código.
El uso puede ser en decir: hemos iniciado aquí pero ahora construiremos algo muy diferente
Vemos dónde iniciamos
ggplot(data = penguins)

Vemos dónde iniciamos
ggplot(data = penguins) + aes(x = flipper_length_mm)

Vemos dónde iniciamos
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity")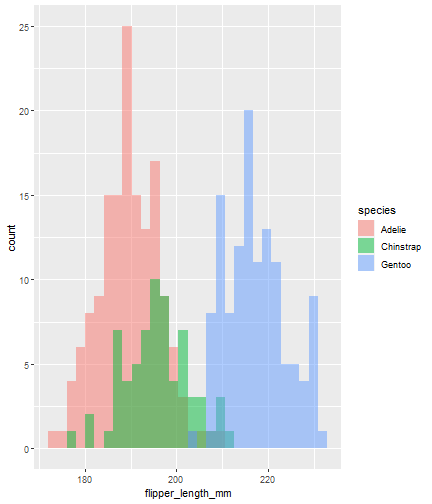

Vemos dónde iniciamos
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal()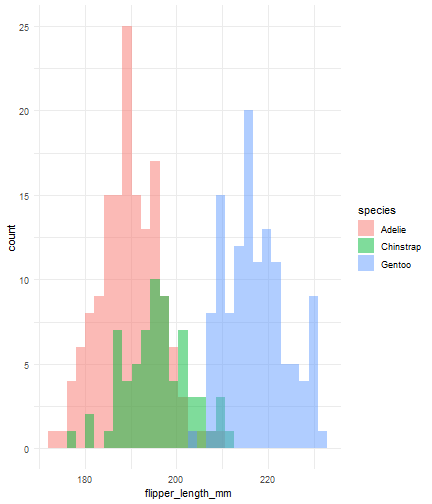

Vemos dónde iniciamos
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal() + labs(x = "Flipper length (mm)", y = "Frequency", title = "Penguin flipper lengths")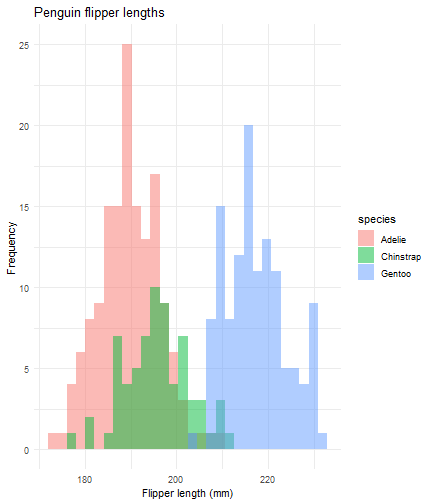

Vemos dónde iniciamos
ggplot(data = penguins) + aes(x = flipper_length_mm) + geom_histogram(aes(fill = species), alpha = 0.5, position = "identity") + theme_minimal() + labs(x = "Flipper length (mm)", y = "Frequency", title = "Penguin flipper lengths") + scale_fill_manual(values = c("darkorange","purple","cyan4"))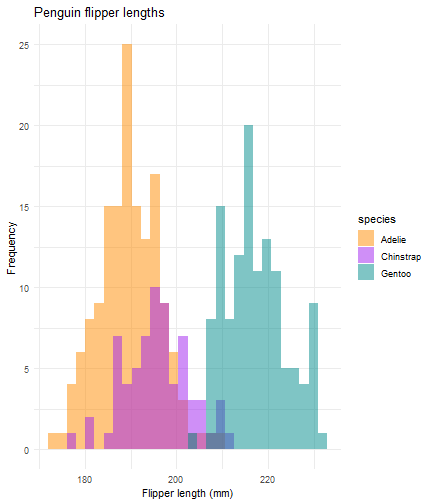

display_type = c("md", "code"), md = c("hi", "by")
Este es el momento...
.. donde podemos incluir texto en los flipbooks. Esto es importante porque posiblmente queremos hacer una breve descripción de lo que pasa en el código o en los resultados.
Para poder "meter texto":
- Agregamos "md" al vector asignado a
display_type - Agregamos otro parámetro a la función
chunk_reveal()que se llame md. - Asignamos al parámetro
mdel vector de caracteres que queremos que aparezcan en cada click.
display_type = c("md", "code"), md = c("hi", "by")
Este es el momento...
.. donde podemos incluir texto en los flipbooks. Esto es importante porque posiblmente queremos hacer una breve descripción de lo que pasa en el código o en los resultados.
Para poder "meter texto":
- Agregamos "md" al vector asignado a
display_type - Agregamos otro parámetro a la función
chunk_reveal()que se llame md. - Asignamos al parámetro
mdel vector de caracteres que queremos que aparezcan en cada click.
Lo que puede ser complicado es ver cuántos son los elementos que conformarán al vector md. Esto dependerá del break_type que utilices.
Dentro de los caracteres que aparecerán, se pueden agregar también fórmulas con sintáxis de LaTeX.
Es muy útil si se desea explicar código.
Elegir la base
penguinsLlamar al ggplot
penguins %>% ggplot()Elegir el eje x
penguins %>% ggplot() + aes(x = bill_length_mm)Elegir el eje y
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)Elegir la agrupación por especies
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)Poner el geométrico de puntos
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)Diferenciar los puntos por especie en color
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)Cambiar forma según la especie
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)Ajustar una curva con modelos lineales
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE).
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)Cambiar el tema de la gráfica
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()Cambiar los colores por grupo
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))Añadir los títulos, subtítulos y nombres de los ejes
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")Acomodar las dimensiones y posición de los títulos
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")display_type = c("md", "md2")
Si queremos en vez de código, una secuencia de caracteres, por ejemplo, fórmula | explicación, esta opción es muy útil.
display_type = c("md", "md2")
Si queremos en vez de código, una secuencia de caracteres, por ejemplo, fórmula | explicación, esta opción es muy útil.
Como en la explicación anterior se requiere:
Un vector "de caracteres o fórmulas" asignado al argumento
mden la funciónchunk_reveal()Un vector "de caracteres o fórmulas" asignado al argumento
md2en la funciónchunk_reveal()display_type = c("md", "md2")en el argumento de la funciónchunk_reveal()
display_type = c("md", "md2")
Si queremos en vez de código, una secuencia de caracteres, por ejemplo, fórmula | explicación, esta opción es muy útil.
Como en la explicación anterior se requiere:
Un vector "de caracteres o fórmulas" asignado al argumento
mden la funciónchunk_reveal()Un vector "de caracteres o fórmulas" asignado al argumento
md2en la funciónchunk_reveal()display_type = c("md", "md2")en el argumento de la funciónchunk_reveal()
Fórmula 1
X1+X2+...+Xnn
Fórmula 2
1nn∑i=1Xi
¿Cuál crees que es mejor?
Son lo mismo, la media muestral.
display_type = "func"
Si queremos usar una función y conocer los parámetros para conformarla (pero al mismo tiempo dar a conocer lo que nos devuelve) solo usamos el "func" (y el argumento "output").
Se puede usar con pipelines lo cual quiere decir una "composición" de funciones sobre un argumento.
display_type = "func"
Si queremos usar una función y conocer los parámetros para conformarla (pero al mismo tiempo dar a conocer lo que nos devuelve) solo usamos el "func" (y el argumento "output").
Se puede usar con pipelines lo cual quiere decir una "composición" de funciones sobre un argumento.
Recuerda que display_type puede tener un vector de tres entradas, así que en el siguiente ejemplo podremos ver el flipbook de display_type = c("func", "code", "output")
Es decir que veremos de qué trata la función que se usa, el código correspondiente en el chunk y el resultado del código.
function (n, mean = 0, sd = 1) .Call(C_rnorm, n, mean, sd)<bytecode: 0x00000000051f0df8><environment: namespace:stats>rnorm(50) [1] -1.53147884 1.75166959 -0.41882860 0.13585317 -0.49435011 1.41166016 [7] 0.03562314 1.92860023 1.53909251 -1.24460649 1.30823105 -0.39640682[13] -0.78292370 -0.52375592 -1.17286749 -0.33130001 -1.29103472 -1.44679700[19] 0.16170908 1.43641497 0.84370559 -0.88190602 1.10546986 -1.02006868[25] 0.73895122 -0.14344446 -0.29824149 0.05437185 -0.39506909 1.89324696[31] 0.29101814 2.04557854 -0.37894783 0.28911027 -0.85036158 -0.54221805[37] -0.41290449 -0.70370288 0.28916620 1.28916097 0.94076266 -0.02061201[43] 0.77557886 -0.66791031 -0.11697377 -0.43013544 0.75358343 0.02381247[49] -0.91984694 0.25370952function (x, na.rm = FALSE) sqrt(var(if (is.vector(x) || is.factor(x)) x else as.double(x), na.rm = na.rm))<bytecode: 0x000000002dfc90a0><environment: namespace:stats>rnorm(50) %>% sd()[1] 1.066501function (x) .Primitive("length")rnorm(50) %>% sd() %>% length()[1] 1Extra extra!
Extra extra!
Para añadir un título al flipbook, simplemente es necesario que escribas dentro de la función
chunk_reveal()el parámetrotitle = "## Mi primer títuloPosiblemente tengas interés en cambiar la forma en que se desarrolla tu flipbook, es decir que a lo mejor quieres que...
Extra extra!
Para añadir un título al flipbook, simplemente es necesario que escribas dentro de la función
chunk_reveal()el parámetrotitle = "## Mi primer títuloPosiblemente tengas interés en cambiar la forma en que se desarrolla tu flipbook, es decir que a lo mejor quieres que...
.. se vea el código encima del resultado.
Esto es posible y está en desarrollo aún, pero se logra con agregar el parámetro float dentro de la función chunk_reveal() con las opciones "top" o "bottom"
Experimenta otras formas de visualizar el flipbook
penguins# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot()
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")
Experimenta otras formas de visualizar el flipbook
penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")
left_type
left_type
Este parámetro de la función chunk_reveal() ayuda a decidir cómo "leer" el código dento del chunk.
Assignment
Para crear objetos en R es compún usar <- pero igual se puede usar -> o incluso =.
El objetivo de usar left_assign = TRUE es darle "prioridad" al objeto de asignación. Esto se nota en que aparece siempre el objeto creado durante el flipbook creado.
Assignment
Para crear objetos en R es compún usar <- pero igual se puede usar -> o incluso =.
El objetivo de usar left_assign = TRUE es darle "prioridad" al objeto de asignación. Esto se nota en que aparece siempre el objeto creado durante el flipbook creado.
La utilidad es que podremos conocer el desarrollo de un objeto de manera paulatina y además ver el resultado final. Y posteriormente modificar ese objeto.
Además sino se usa left_assign = TRUE (que es lo que sucede por defecto) entonces no se podrá hacer este tipo de asignación (->).
pinguinos1 <- penguinspinguinos1# A tibble: 344 x 8 species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g <fct> <fct> <dbl> <dbl> <int> <int> 1 Adelie Torgersen 39.1 18.7 181 3750 2 Adelie Torgersen 39.5 17.4 186 3800 3 Adelie Torgersen 40.3 18 195 3250 4 Adelie Torgersen NA NA NA NA 5 Adelie Torgersen 36.7 19.3 193 3450 6 Adelie Torgersen 39.3 20.6 190 3650 7 Adelie Torgersen 38.9 17.8 181 3625 8 Adelie Torgersen 39.2 19.6 195 4675 9 Adelie Torgersen 34.1 18.1 193 347510 Adelie Torgersen 42 20.2 190 4250# ... with 334 more rows, and 2 more variables: sex <fct>, year <int>pinguinos1 <- penguins %>% ggplot()pinguinos1
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm)pinguinos1
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm)pinguinos1
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species)pinguinos1
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8)pinguinos1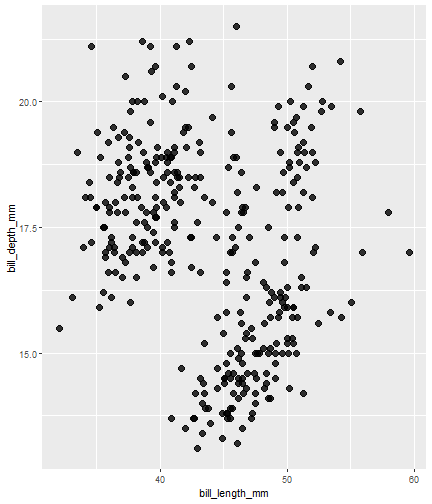
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species)pinguinos1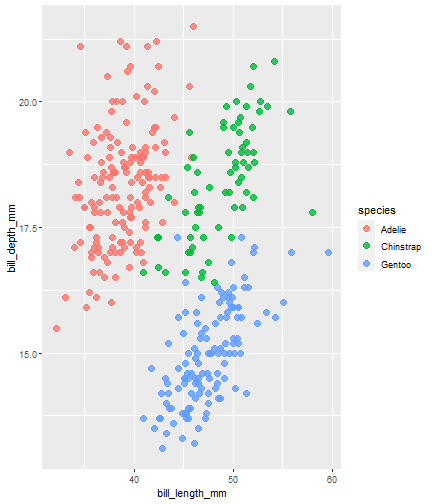
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species)pinguinos1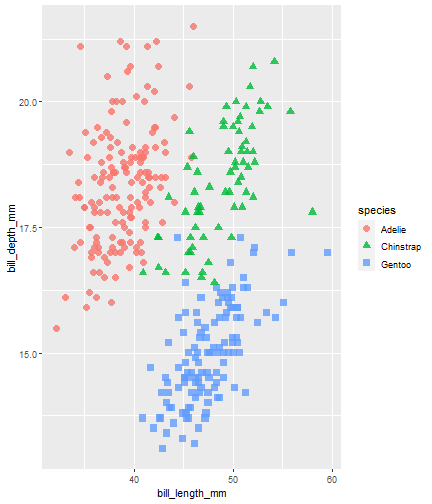
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE)pinguinos1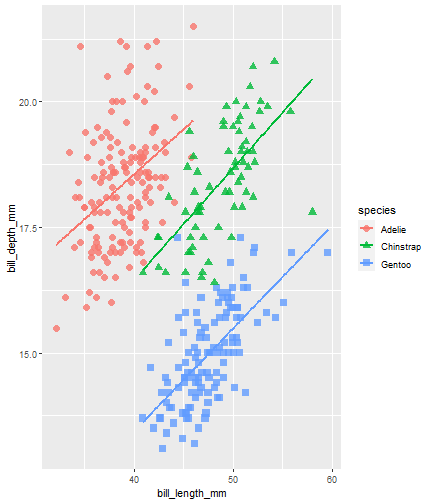
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species)pinguinos1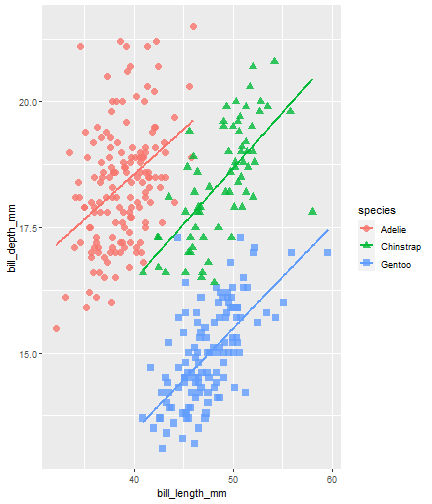
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal()pinguinos1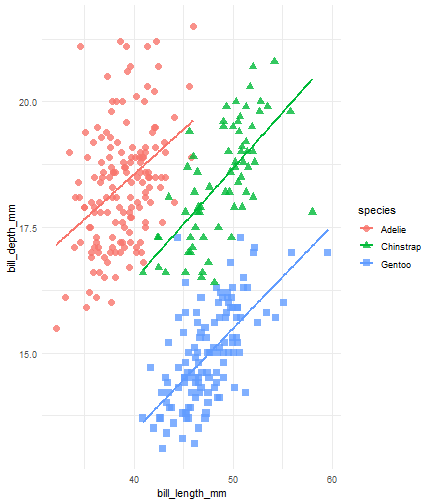
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17"))pinguinos1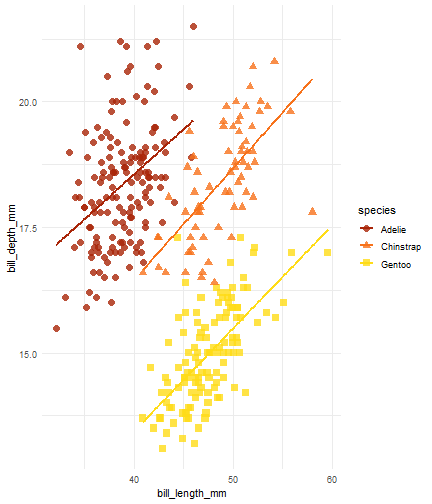
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino")pinguinos1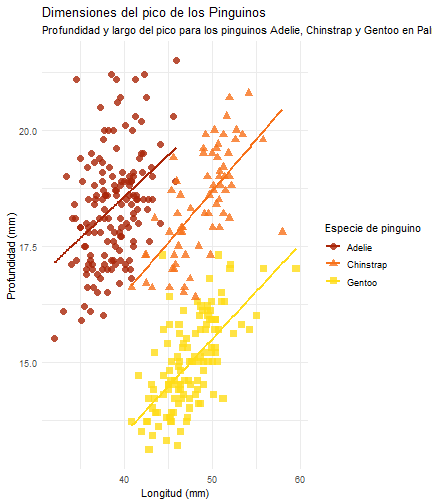
pinguinos1 <- penguins %>% ggplot() + aes(x = bill_length_mm) + aes(y = bill_depth_mm) + aes(group = species) + geom_point(size = 3, alpha = 0.8) + aes(color = species) + aes(shape = species) + geom_smooth(method = "lm", se = FALSE) + aes(color = species) + theme_minimal() + scale_color_manual(values = c("#A92508", "#F7721B", "#FFDC17")) + labs(title = "Dimensiones del pico de los Pinguinos", subtitle = "Profundidad y largo del pico para los pinguinos Adelie, Chinstrap y Gentoo en Palmer Station LTER", x = "Longitud (mm)", y = "Profundidad (mm)", color = "Especie de pinguino", shape = "Especie de pinguino") + theme( legend.position = c(0.85, 0.15), legend.background = element_rect(fill = "white", color = NA), plot.title.position = "plot", plot.caption = element_text(hjust = 0, face = "italic"), plot.caption.position = "plot")pinguinos1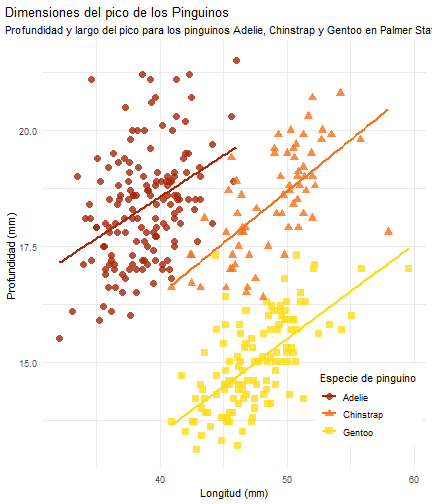
Más allá de gráficas y datos
Más allá de gráficas y datos
Podemos hacer flipbooks de cualquier cosa.
Operaciones aritméticas
Hacer operaciones aritméticas de forma paulatina tambipen es posible pero... como sabes, debe seguirse la jerarquía de operaciones y no solo por orden de aparición en el chunk de código.
Operaciones aritméticas
Hacer operaciones aritméticas de forma paulatina tambipen es posible pero... como sabes, debe seguirse la jerarquía de operaciones y no solo por orden de aparición en el chunk de código.

(4 + 5) # Hay que tener cuidado[1] 9(4 + 5) / # Hay que tener cuidado 6 # porque el orden de las operaciones[1] 1.5(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 # considera[1] 10.5(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 - # considera 3 # el orden jerárquico[1] 7.5(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 - # considera 3 * # el orden jerárquico 12 # de Matemáticas[1] -25.5(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 - # considera 3 * # el orden jerárquico 12 # de Matemáticasmatrix(1:4, ncol = 1)[1] -25.5 [,1][1,] 1[2,] 2[3,] 3[4,] 4(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 - # considera 3 * # el orden jerárquico 12 # de Matemáticasmatrix(1:4, ncol = 1) %*% matrix(1:4, nrow = 1)[1] -25.5 [,1] [,2] [,3] [,4][1,] 1 2 3 4[2,] 2 4 6 8[3,] 3 6 9 12[4,] 4 8 12 16(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 - # considera 3 * # el orden jerárquico 12 # de Matemáticasmatrix(1:4, ncol = 1) %*% matrix(1:4, nrow = 1)matrix(1:4, ncol = 4)[1] -25.5 [,1] [,2] [,3] [,4][1,] 1 2 3 4[2,] 2 4 6 8[3,] 3 6 9 12[4,] 4 8 12 16 [,1] [,2] [,3] [,4][1,] 1 2 3 4(4 + 5) / # Hay que tener cuidado 6 * # porque el orden de las operaciones 7 - # considera 3 * # el orden jerárquico 12 # de Matemáticasmatrix(1:4, ncol = 1) %*% matrix(1:4, nrow = 1)matrix(1:4, ncol = 4) %*% matrix(1:4, nrow = 4)[1] -25.5 [,1] [,2] [,3] [,4][1,] 1 2 3 4[2,] 2 4 6 8[3,] 3 6 9 12[4,] 4 8 12 16 [,1][1,] 30Hacer flipbook's DE TEXTO
Hacer flipbook's DE TEXTO
text_reveal
Hacer flipbook's DE TEXTO
text_reveal
Es LA FUNCIÓN que sirve para ello
Hacer flipbook's DE TEXTO
text_reveal
Es LA FUNCIÓN que sirve para ello
Dale click de nuevo
Hacer flipbook's DE TEXTO
text_reveal
Es LA FUNCIÓN que sirve para ello
Dale click de nuevo
Conócela
Hacer flipbook's DE TEXTO
text_reveal
Es LA FUNCIÓN que sirve para ello
Dale click de nuevo
Conócela
La entrada es un conjunto de caracteres, y el parámetro sep permitirá que la función sepa CUANDO se harán los "cortes" de la cadena de caracteres.